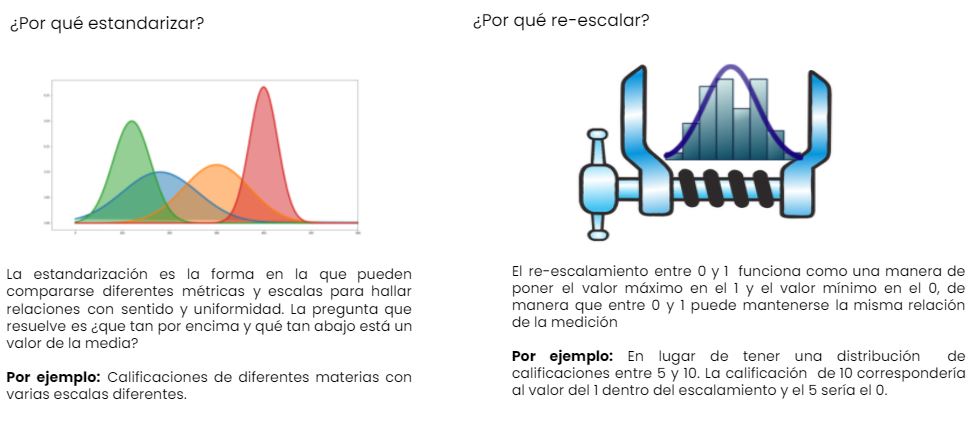
Capítulo 4 MACHINE LEARNING: CONCEPTOS BÁSICOS
4.1 Conocimiento previo de los datos: EDA y GEDA

“El análisis exploratorio de datos se refiere al proceso crítico de realizar investigaciones iniciales sobre los datos para descubrir patrones, detectar anomalías, probar hipótesis y verificar suposiciones con la ayuda de estadísticas resumidas y representaciones gráficas.” Towards

4.1.1 EDA: Análisis Exploratorio de Datos
Un analisis explorario de datos tiene principalmente 5 objetivos:
- Maximizar el conocimiento de un conjunto de datos
- Descubrir la estructura subyacente de los datos
- Extraer variables importantes
- Detectar valores atípicos y anomalías
- Probar los supuestos subyacentes
EDA no es idéntico a los gráficos estadísticos aunque los dos términos se utilizan casi indistintamente. Los gráficos estadísticos son una colección de técnicas, todas basadas en gráficos y todas centradas en un aspecto de caracterización de datos. EDA abarca un lugar más grande. EDA es una filosofía sobre cómo diseccionar un conjunto de datos; lo que buscamos; cómo nos vemos; y cómo interpretamos.
Los científicos de datos pueden utilizar el análisis exploratorio para garantizar que los resultados que producen sean válidos y aplicables a los resultados y objetivos comerciales deseados.
EDA se utiliza principalmente para ver qué datos pueden revelar más allá del modelado formal o la tarea de prueba de hipótesis y proporciona una mejor comprensión de las variables del conjunto de datos y las relaciones entre ellas. También puede ayudar a determinar si las técnicas estadísticas que está considerando para el análisis de datos son apropiadas.
Dependiendo del tipo de variable queremos obtener la siguiente información:
Variables numéricas:
- Tipo de dato: float, integer
- Número de observaciones
- Mean
- Desviación estándar
- Cuartiles: 25%, 50%, 75%
- Valor máximo
- Valor mínimo
- Número de observaciones únicos
- Top 5 observaciones repetidas
- Número de observaciones con valores faltantes
- ¿Hay redondeos?
Variables categóricas
- Número de categorías
- Valor de las categorías
- Moda
- Valores faltantes
- Número de observaciones con valores faltantes
- Proporción de observaciones por categoría
- Top 1, top 2, top 3 (moda 1, moda 2, moda 3)
- Faltas de ortografía ?
Fechas
- Fecha inicio
- Fecha fin
- Huecos en las fechas: sólo tenemos datos entre semana, etc.
- Formatos de fecha (YYYY-MM-DD)
- Tipo de dato: date, time, timestamp
- Número de faltantes (NA)
- Número de observaciones
Texto
- Longitud promedio de cada observación
- Identificar el lenguaje, si es posible
- Longitud mínima de cada observación
- Longitud máxima de cada observación
- Cuartiles de longitud: 25%, 50%, 75%
Coordenadas geoespaciales
- Primero se pone la latitud y luego la longitud
- Primer decimal: 111 kms
- Segundo decimal: 11.1 kms
- Tercer decimal: 1.1 kms
- Cuarto decimal: 11 mts
- Quinto decimal: 1.1 mt
- Sexto decimal: 0.11 mts
- Valores que están cercanos al 100 representan la longitud
- El símbolo en cada coordenada representa si estamos al norte (positivo) o sur (negativo) -en la latitud-, al este (positivo) o al - oeste (negativo) -en la longitud-.
4.1.2 GEDA: Análisis Exploratorio de Datos Gráficos
Como complemento al EDA podemos realizar un GEDA, que es un análisis exploratorio de los datos apoyándonos de visualizaciones, la visualización de datos no trata de hacer gráficas “bonitas” o “divertidas,” ni de simplificar lo complejo. Más bien, trata de aprovechar nuestra gran capacidad de procesamiento visual para exhibir de manera clara aspectos importantes de los datos.
4.1.2.1 Lo que no se debe hacer…

Fuentes: WTF Visualizations Flowingdata
{r echo=FALSE,fig.align=‘center,’ out.width = “700pt”} knitr::include_graphics(“img/03-viz/bad_viz3.png”)

4.1.2.2 Principios de visualización
El objetivo de una visualización es sintentizar información relevante al análisis presentada de manera sencilla y sin ambigüedad. Lo usamos de apoyo para explicar a una audiencia más amplia que puede no ser tan técnica.
Una gráfica debe reportar el resultado de un análisis detallado, nunca lo reemplaza.
No hacer gráficas porque se vean “cool”
Antes de hacer una gráfica, debe pensarse en lo que se quiere expresar o representar
Existen “reglas” o mejores gráficas para representar cierto tipo de información de acuerdo a los tipos de datos que se tienen o al objetivo se quiere lograr con la visualización. From Data to Viz
No utilizar pie charts
4.1.2.3 Principios generales del diseño analítico:
Muestra comparaciones, contrastes, diferencias.
Muestra causalidad, mecanismo, explicación.
Muestra datos multivariados, es decir, más de una o dos variables.
Integra palabras, números, imágenes y diagramas.
Las presentaciones analíticas, a fin de cuentas, se sostienen o caen dependiendo de la calidad, relevancia e integridad de su contenido.
Esta categoría incluye técnicas específicas que dependen de la forma de nuestros datos y el tipo de pregunta que queremos investigar:
4.1.2.4 Técnicas de visualización:
Tipos de gráficas: cuantiles, histogramas, caja y brazos, gráficas de dispersión, puntos/barras/ líneas, series de tiempo.
Técnicas para mejorar gráficas: Transformación de datos, transparencia, vibración, suavizamiento y bandas de confianza.
4.1.2.5 Indicadores de calidad gráfica:
Aplicables a cualquier gráfica en particular, son guías concretas y relativamente objetivas para evaluar la calidad de una gráfica.
Integridad Gráfica: El factor de engaño, es decir, la distorsión gráfica de las cantidades representadas, debe ser mínimo.
Chartjunk: Minimizar el uso de decoración gráfica que interfiera con la interpretación de los datos: 3D, rejillas, rellenos con patrones.
Tinta de datos: Maximizar la proporción de tinta de datos vs. tinta total de la gráfica. La regla es: si hay tinta que no representa variación en los datos, o la eliminación de esa tinta no representa pérdidas de significado, esa tinta debe ser eliminada. El ejemplo más claro es el de las rejillas en gráficas y tablas:
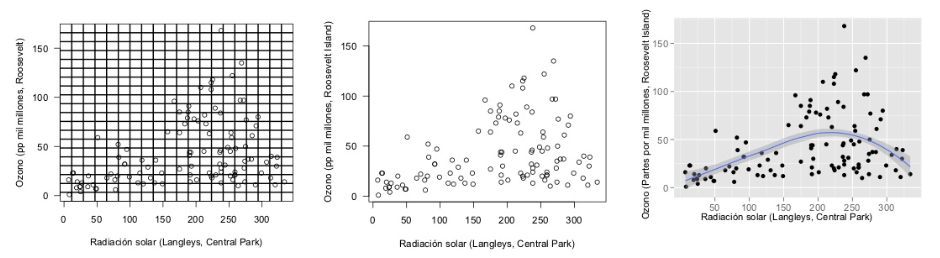
- Densidad de datos: Las mejores gráficas tienen mayor densidad de datos, que es la razón entre el tamaño del conjunto de datos y el área de la gráfica.
4.1.3 Uso, decisión e implementación de técnicas gráficas.
4.1.3.1 Gráficos univariados:
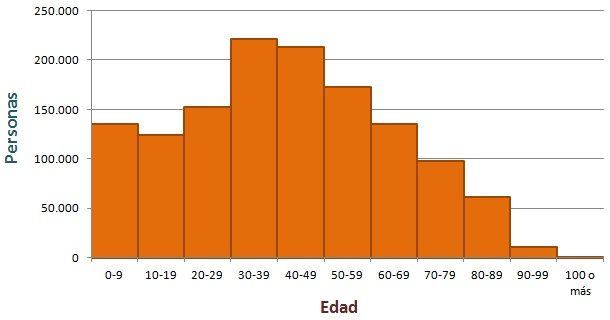
- Histograma: El histograma es la forma más popular de mostrar la forma de un conjunto de datos. Se divide la escala de la variable en intervalos, y se realiza un conteo de los casos que caen en cada uno de los intervalos. Los histogramas pueden mostrar distintos aspectos de los datos dependiendo del tamaño y posición de los intervalos.
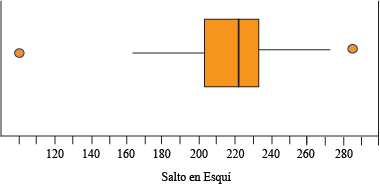
- Diagramas de caja y brazos: Es un método estandarizado para representar gráficamente una serie de datos numéricos a través de sus cuartiles. El diagrama de caja muestra a simple vista la mediana y los cuartiles de los datos, pudiendo también representar los valores atípicos de estos.
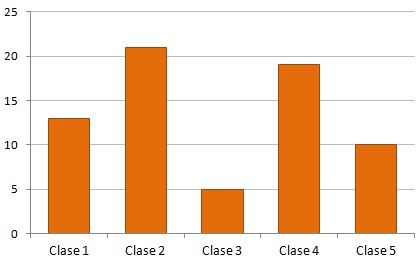
- Gráficas de barras: Una gráfica de este tipo nos muestra la frecuencia con la que se han observado los datos de una variable discreta, con una barra para cada categoría de esta variable.
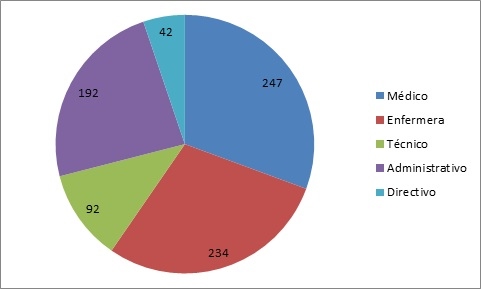
- Gráficos Circulares (Pie Charts): Un gráfico circular o gráfica circular, también llamado “gráfico de pastel,” es un recurso estadístico que se utiliza para representar porcentajes y proporciones.
4.1.3.2 Gráficos multivariados
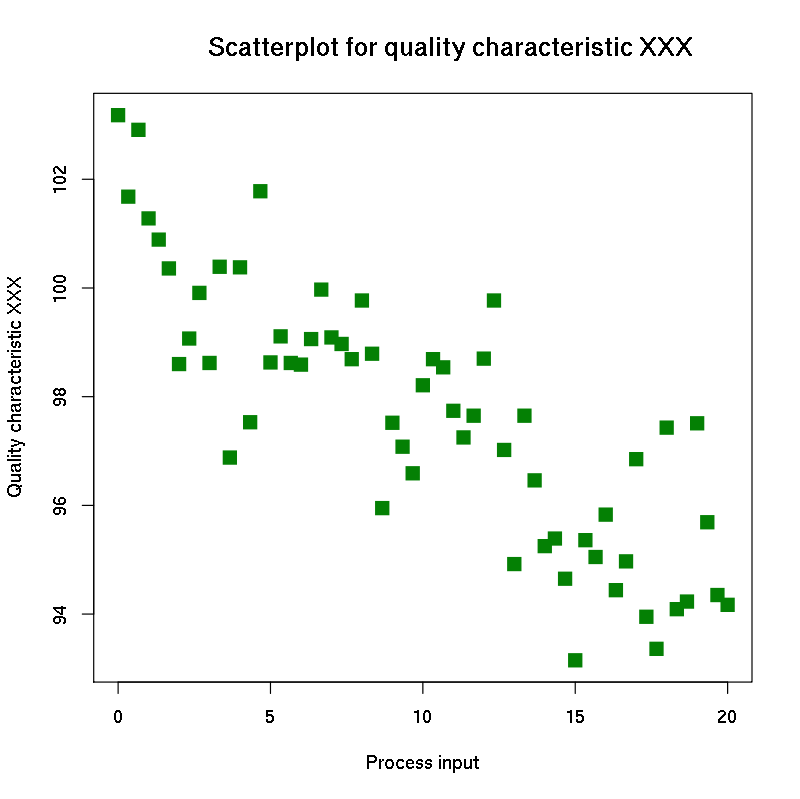
- Gráfico de dispersión: Los gráficos de dispersión se usan para trazar puntos de datos en un eje vertical y uno horizontal, mediante lo que se trata de mostrar cuánto afecta una variable a otra. Si no existe una variable dependiente, cualquier variable se puede representar en cada eje y el diagrama de dispersión mostrará el grado de correlación (no causalidad) entre las dos variables.
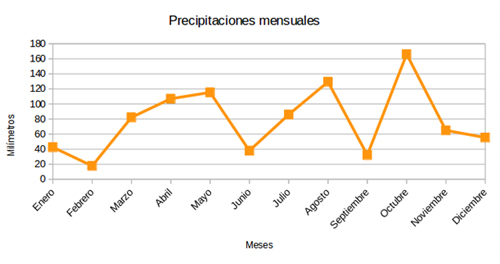
- Gráficas de líneas: Uno de los tipos de gráfica más utilizados es la de líneas, especialmente cuando se quieren comparar visualmente varias variables a lo largo del tiempo o algún otro parámetro.
4.1.4 Ggplot2
Comparando con los gráficos base de R, ggplot2:
- Tiene una gramática más compleja para gráficos simples
- Tiene una gramática menos compleja para gráficos complejos o muy customizados.
- Los datos siempre deben ser un data.frame.
- Usa un sistema diferente para añadir elementos al gráfico.
Histograma con los gráficos base:
data(iris)
hist(iris$Sepal.Length)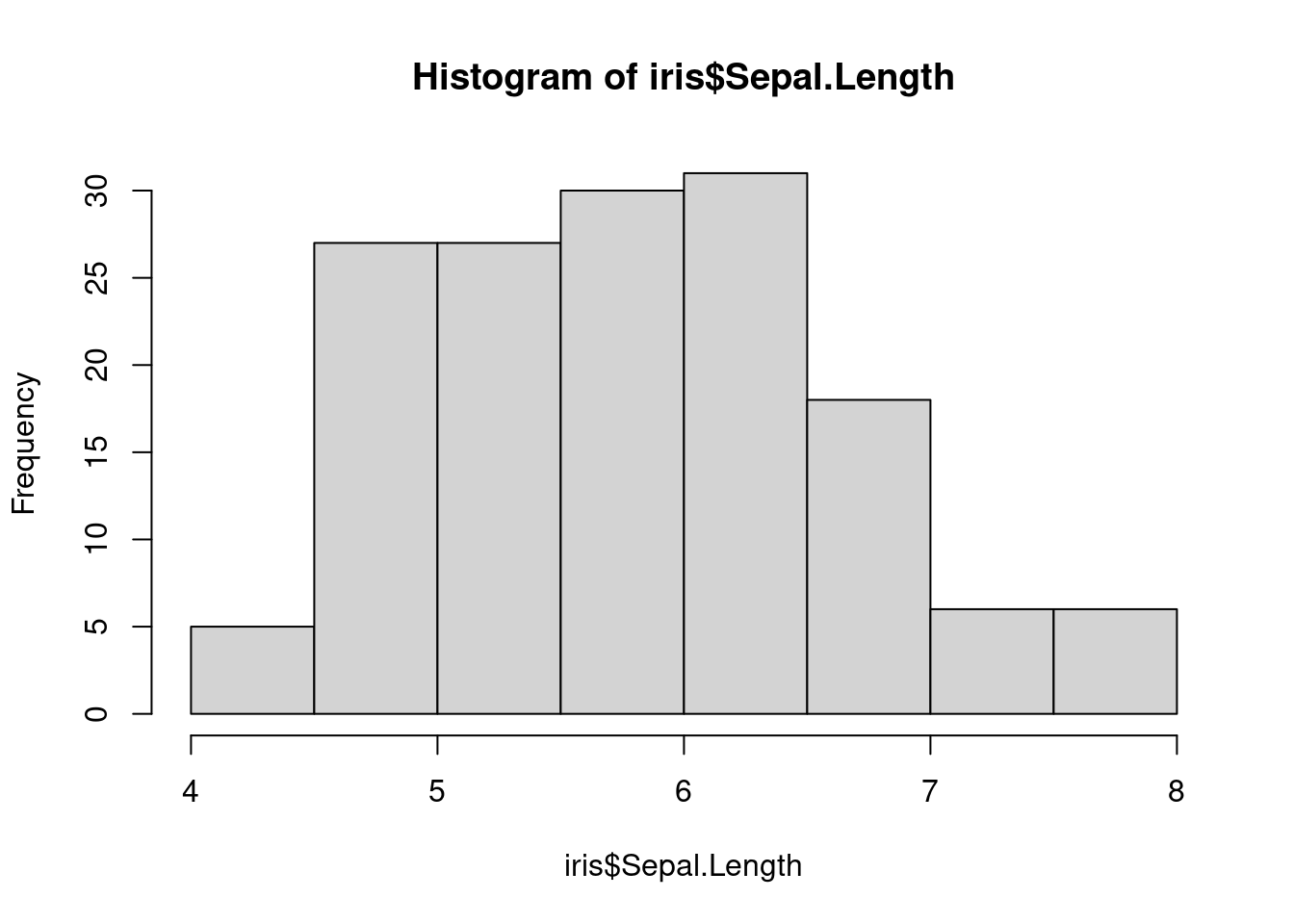
Histograma con ggplot2:
library(ggplot2)
ggplot(iris, aes(x = Sepal.Length)) +
geom_histogram(color = 'white', bins=8)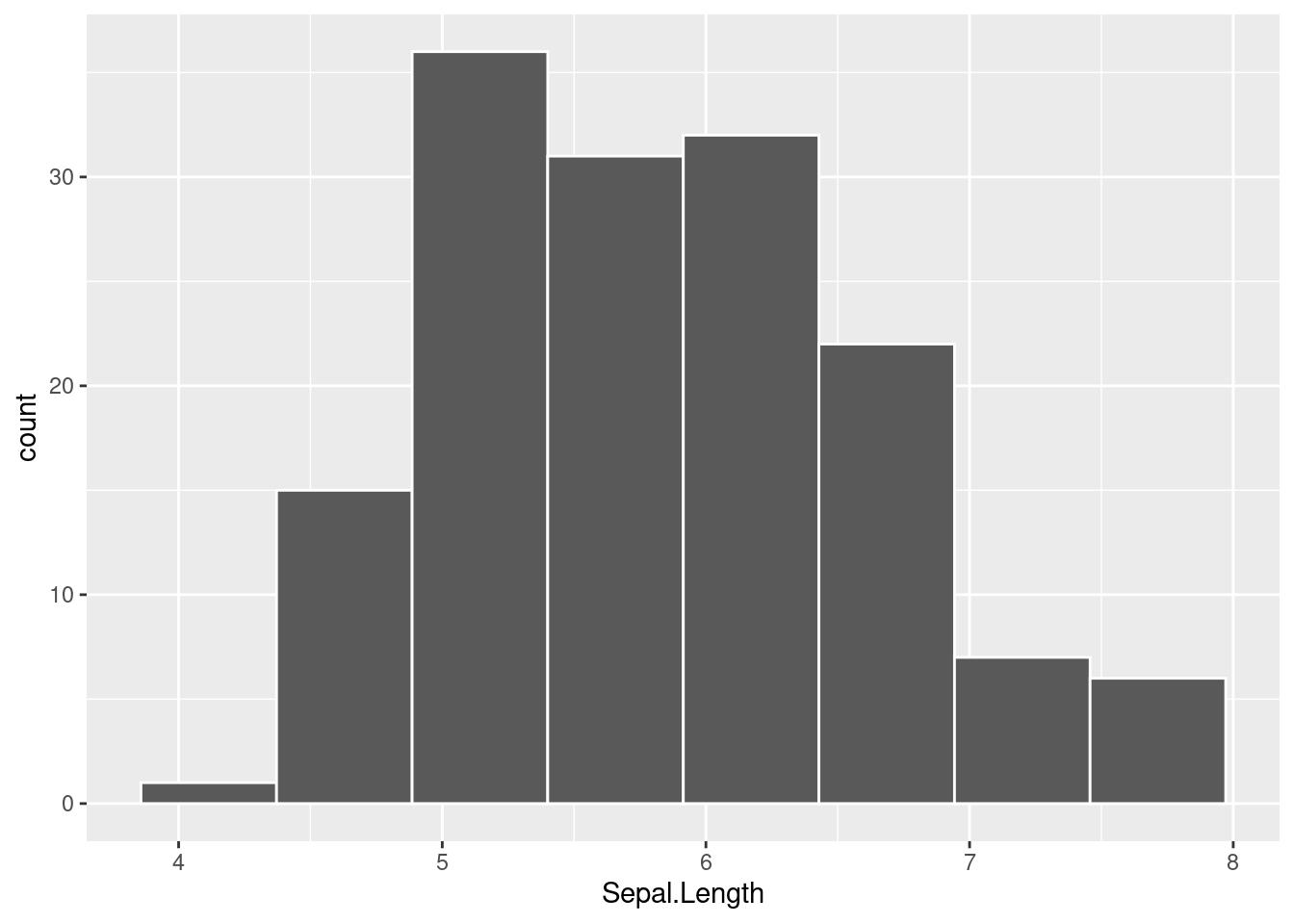
Ahora vamos a ver un gráfico con colores y varias series de datos.
Con los gráficos base:
plot(Sepal.Length ~ Sepal.Width,
col = factor(Species),
data = iris)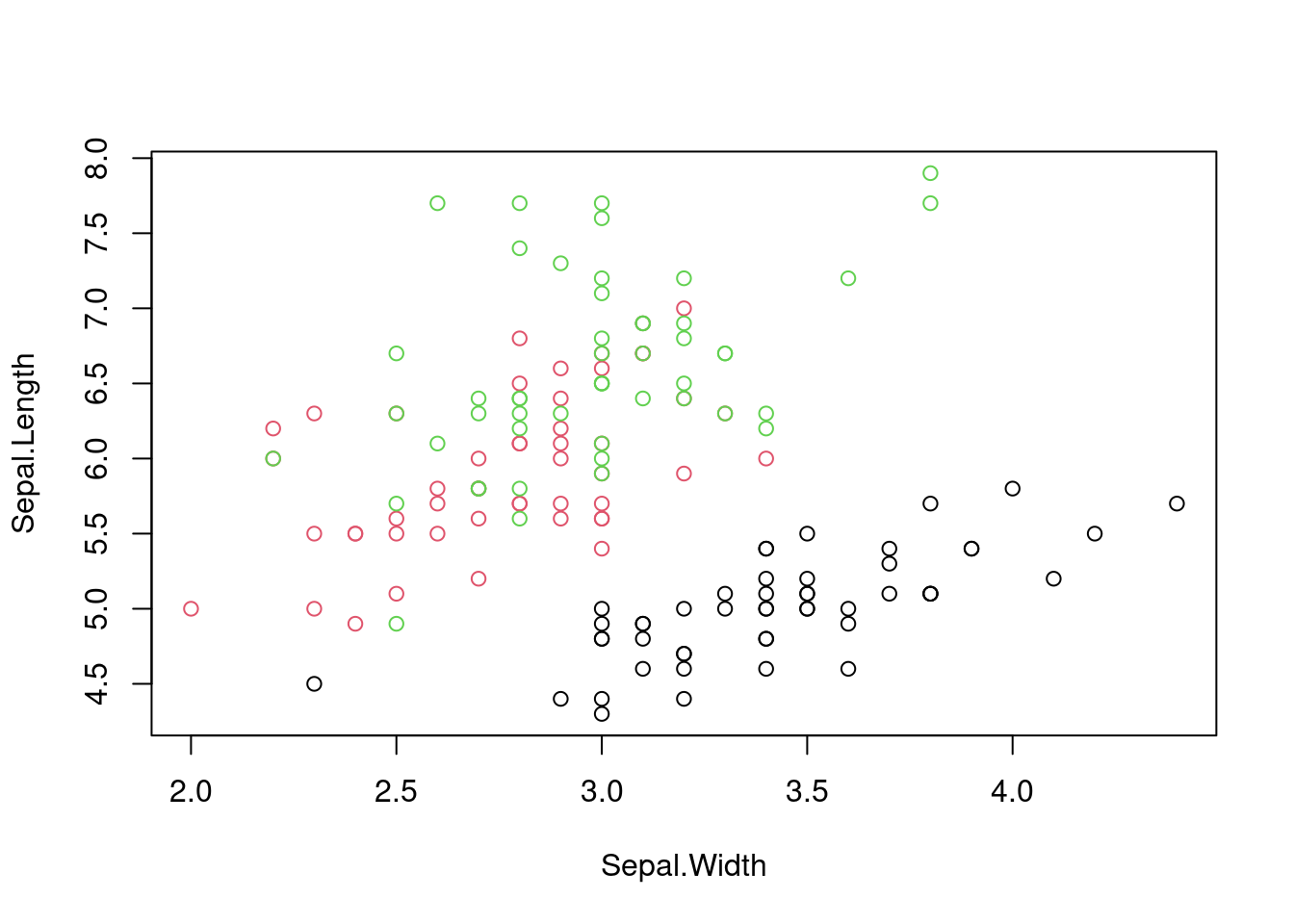
Con ggplot2:
ggplot(iris,
aes(x=Sepal.Width ,
y= Sepal.Length,
color=Species))+
geom_point()
4.1.4.1 Objetos aesteticos
En ggplot2, aestético significa “algo que puedes ver.”
Algunos ejemplos son:
- Posición (por ejemplo, los ejes x e y)
- Color (color “externo”)
- Fill (color de relleno)
- Shape (forma de puntos)
- Linetype (tipo de linea)
- Size (tamaño)
- Alpha (para la transparencia: los valores más altos tendrían formas opacas y los más bajos, casi transparentes).
Hay que advertir que no todas las estéticas tienen la misma potencia en un gráfico. El ojo humano percibe fácilmente longitudes distintas. Pero tiene problemas para comparar áreas (que es lo que regula la estética size) o intensidades de color. Se recomienda usar las estéticas más potentes para representar las variables más importantes.
Cada tipo de objeto geométrico (geom) solo acepta un subconjunto de todos los aestéticos. Puedes consultar la pagina de ayuda de geom() para ver que aestéticos acepta. El mapeo aestético se hace con la función aes().
4.1.4.2 Objetos geométricos o capas
Los objetos geométricos son las formas que puede tomar un gráfico. Algunos ejemplos son:
- Barras (geom_bar(), para las variables univariados discretos o nominales)
- Histogramas (geom_hist() para aquellas variables univariadas continuas)
- Puntos (geom_point() para scatter plots, gráficos de puntos, etc…)
- Lineas (geom_line() para series temporales, lineas de tendencia, etc…)
- Cajas (geom_boxplot() para gráficos de cajas)
- Un gráfico debe tener al menos un geom, pero no hay limite. Puedes añadir más geom usando el signo +.
Si queremos conocer la lista de objetos geométricos podemos ejercutar el siguiente código:
help.search("geom_", package = "ggplot2")Una vez añadida una capa al gráfico a este pueden agregarse nuevas capaas
ggplot(iris, aes(x = Petal.Length, y = Petal.Width, colour = Species)) +
geom_point()
ggplot(iris, aes(x = Petal.Length, y = Petal.Width, colour = Species)) +
geom_point()+
geom_smooth()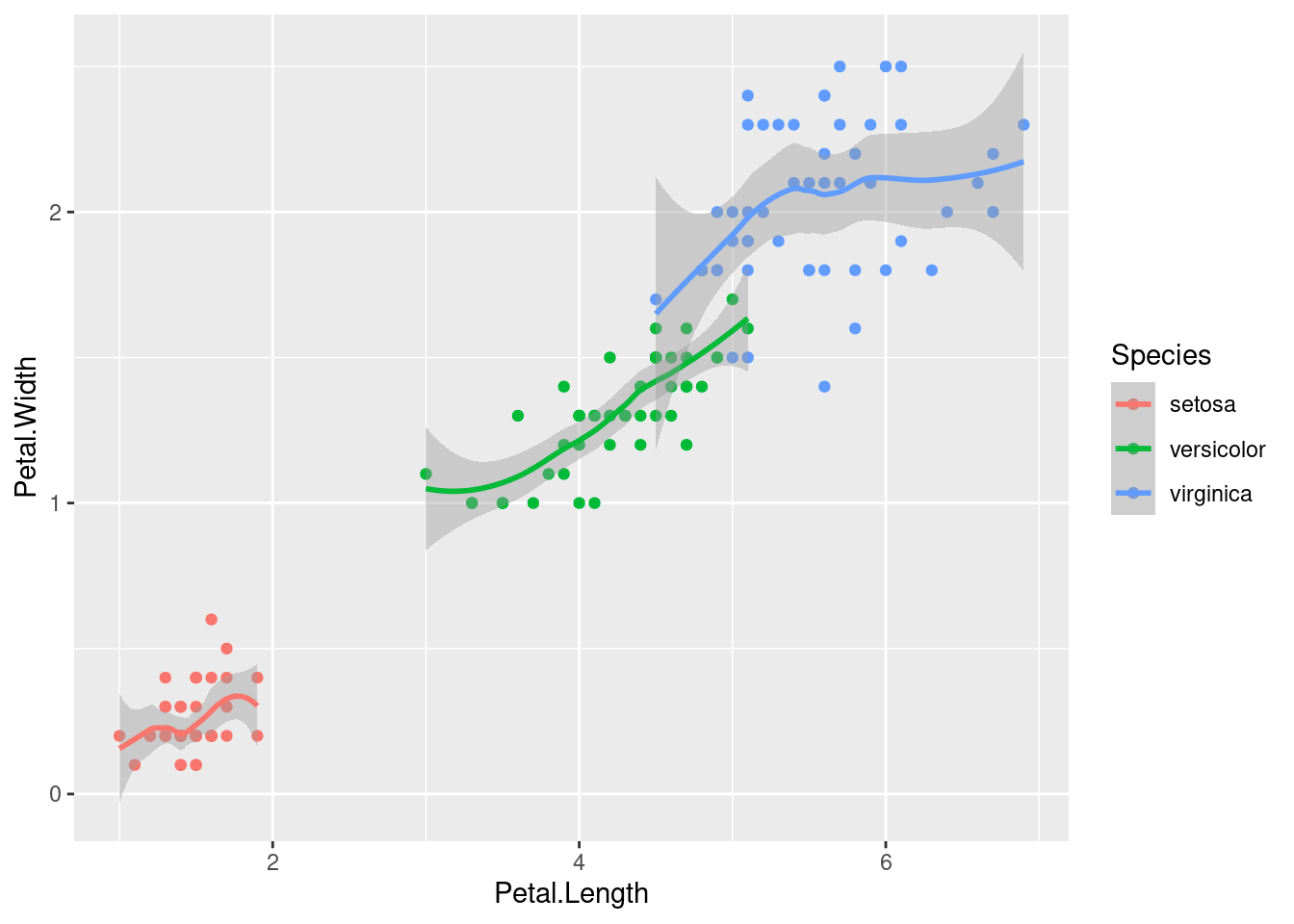
4.1.4.3 Facetas
Muchos de los gráficos que pueden generarse con los elementos anteriores pueden reproducirse usando los gráficos tradicionales de R, pero no los que usan facetas, que pueden permitirnos explorar las variables de diferente forma, por ejemplo:
ggplot(iris, aes(x = Petal.Length, y = Petal.Width)) +
geom_point() + geom_smooth() +
facet_grid(~ Species)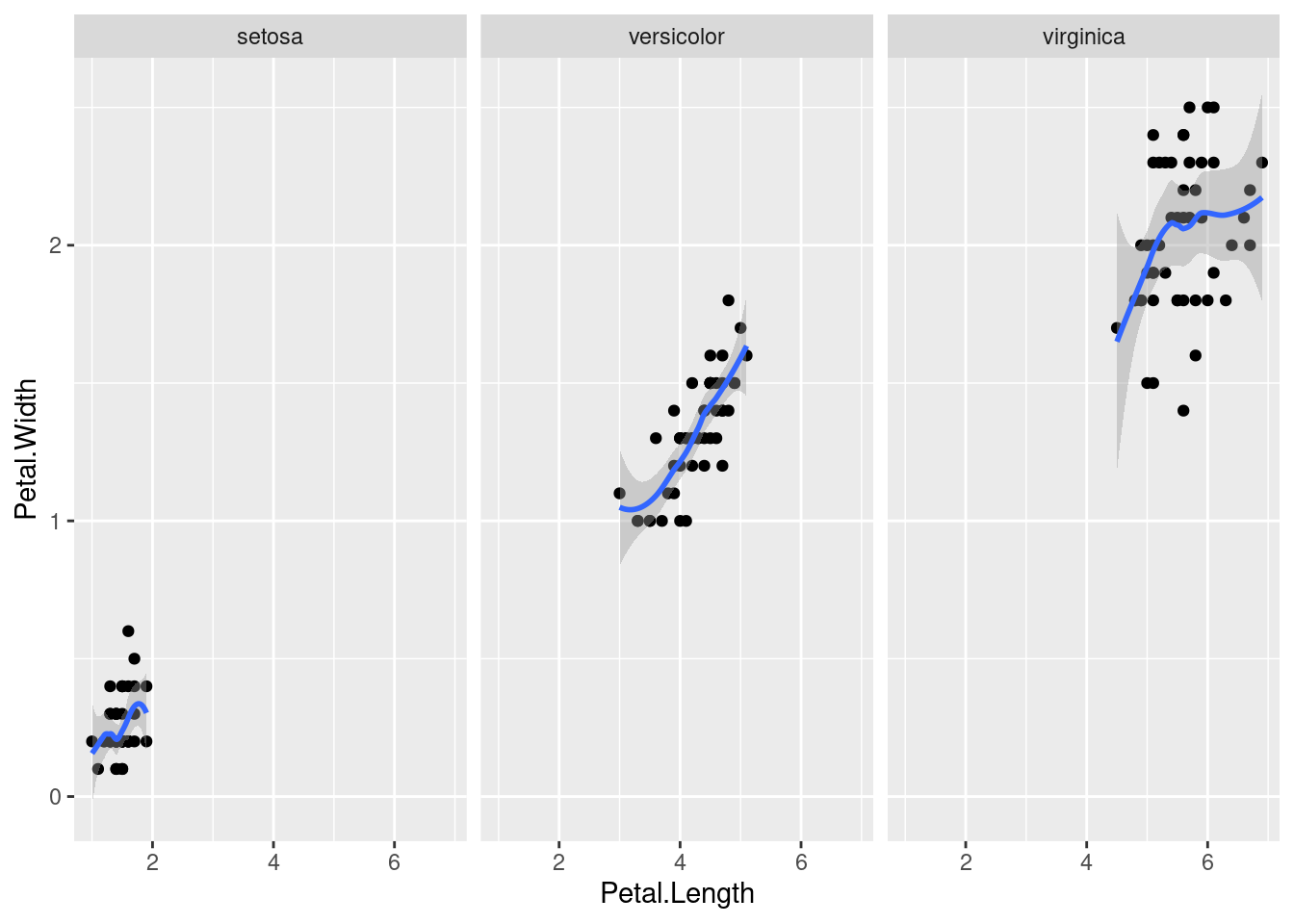
crea tres gráficos dispuestos horizontalmente que comparan la relación entre la anchura y la longitud del pétalo de las tres especies de iris. Una característica de estos gráficos, que es crítica para poder hacer comparaciones adecuadas, es que comparten ejes.
4.1.4.4 Más sobre estéticas
Las estéticas se pueden etiquetar con la función labs. Además, se le puede añadir un título al gráfico usando la función ggtitle. Por ejemplo, en el gráfico anterior se pueden reetiquetar los ejes y la leyenda haciendo
4.1.5 EDA y GEDA con R
Para los ejercicios en clase utilizaremos el set de datos: Diamonds:
library(dplyr)
library(ggplot2)
library(reshape2)
data("diamonds")Descripción Un conjunto de datos que contiene los precios y otros atributos de casi 54.000 diamantes. Las variables son las siguientes:
- price: precio en dólares estadounidenses ( $ 326 - $ 18,823)
- carat: peso del diamante (0.2–5.01)
- cut: calidad del corte (Regular, Bueno, Muy Bueno, Premium, Ideal)
- color: color del diamante, de D (mejor) a J (peor)
- clarity: una medida de la claridad del diamante (I1 (peor), SI2, SI1, VS2, VS1, VVS2, VVS1, IF (mejor))
- x: longitud en mm (0-10,74)
- y: ancho en mm (0–58,9) width in mm (0–58.9) -z: profundidad en mm (0–31,8)
- depth porcentaje de profundidad total = z / media (x, y) = 2 * z / (x + y) (43–79)
- table: ancho de la parte superior del diamante en relación con el punto más ancho (43–95)
Ejemplo práctico:
diamonds %>%
ggplot(aes(x = cut_number(carat, 5), y = price, color = cut)) +
geom_boxplot() +
labs(title = "Distribución de precio por categoría de corte") +
labs (caption = "Data source:Diamont set") +
labs(x = "Peso del diamante") +
labs(y = "Precio")+
guides(color=guide_legend(title="Calidad del corte")) +
ylim(0, 20000) +
scale_y_continuous(labels = scales::dollar_format(),
breaks = seq(0, 20000,2500 ),
limits = c(0, 20000))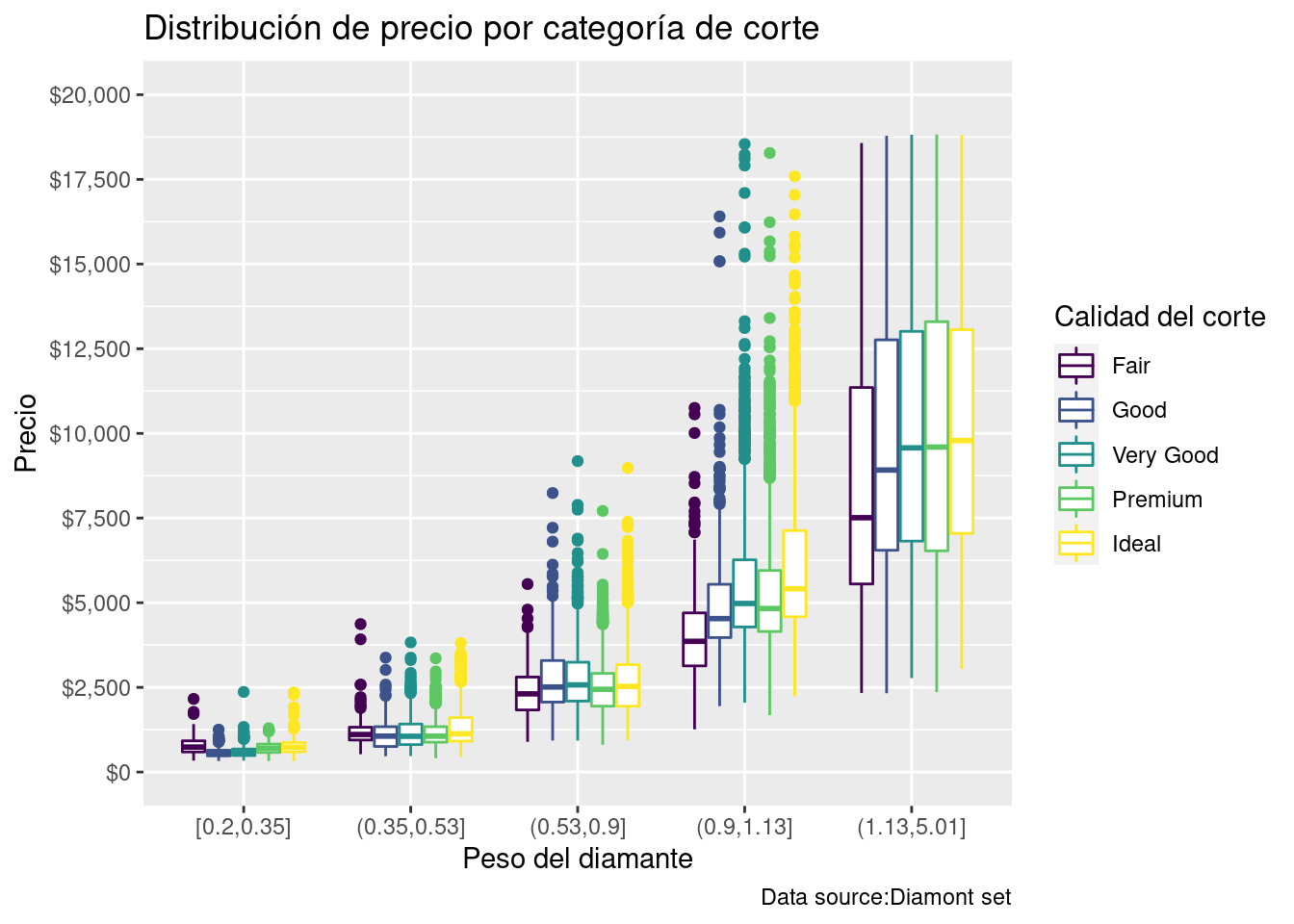
4.1.5.1 Un vistazo rápido a los datos
library(DataExplorer)
plot_intro(diamonds)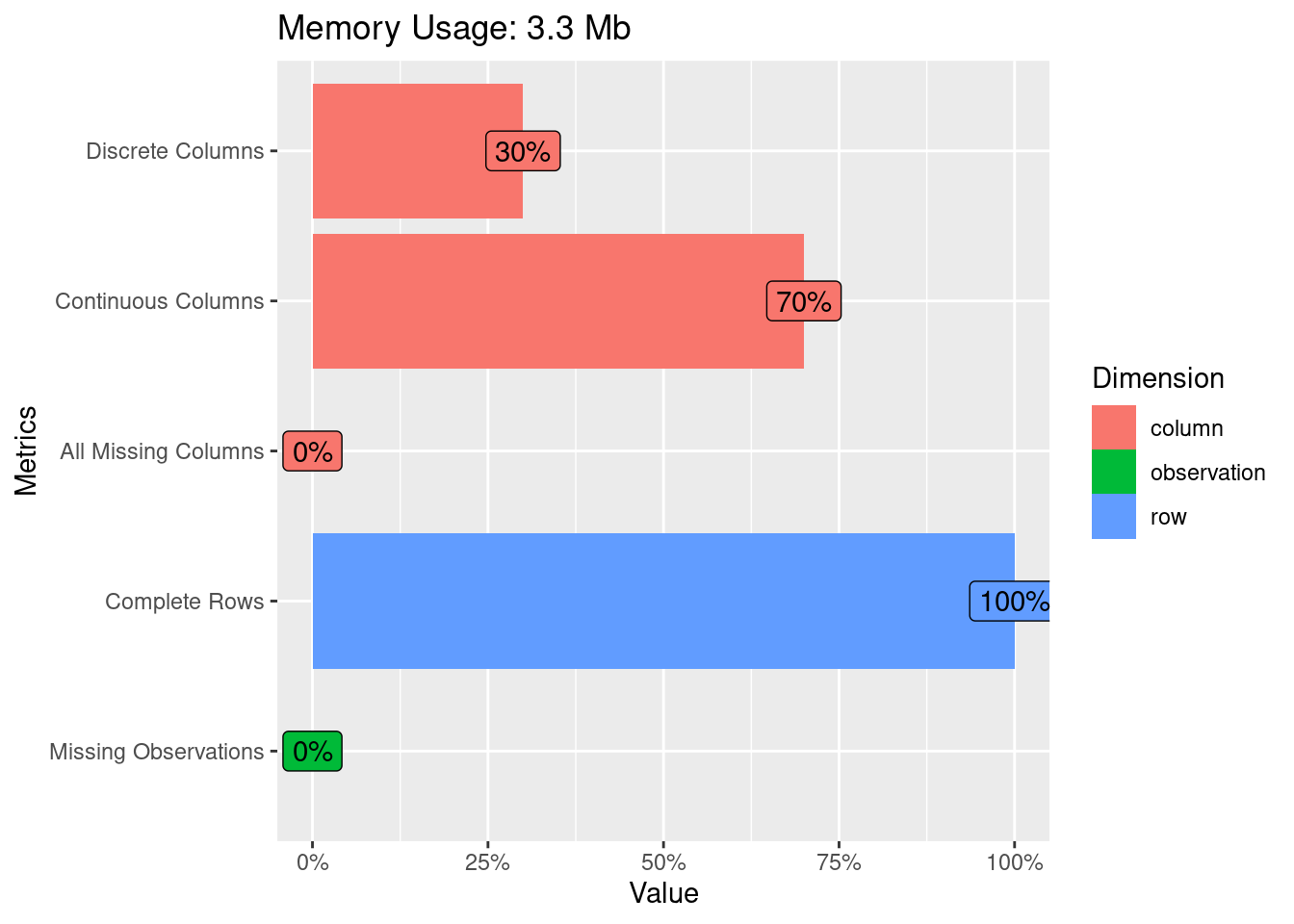
plot_missing(diamonds)
4.1.5.2 Análisis univariado
4.1.5.3 Variables numéricas
Los histogramas son gráficas de barras que se obtienen a partir de tablas de frecuencias, donde cada barra se escala según la frecuencia relativa entre el ancho del intervalo de clase correspondiente.
Un histograma muestra la acumulación ó tendencia, la variabilidad o dispersión y la forma de la distribución.
El Diagrama de Caja y bigoteses un tipo de gráfico que muestra un resumen de una gran cantidad de datos en cinco medidas descriptivas, además de intuir su morfología y simetría.
Este tipo de gráficos nos permite identificar valores atípicos y comparar distribuciones. Además de conocer de una forma cómoda y rápida como el 50% de los valores centrales se distribuyen.
Se puede detectar rápidamente los siguientes valores:
Primer cuartil: el 25% de los valores son menores o igual a este valor (punto 2 en el gráfico anterior).
Mediana o Segundo Cuartil: Divide en dos partes iguales la distribución. De forma que el 50% de los valores son menores o igual a este valor (punto 3 en el gráfico siguiente).
Tercer cuartil: el 75% de los valores son menores o igual a este valor (punto 4 en el gráfico siguiente).
Rango Intercuartílico (RIC): Diferencia entre el valor del tercer cuartil y el primer cuartil.
Tip: El segmento que divide la caja en dos partes es la mediana (punto 3 del gráfico), que facilitará la comprensión de si la distribución es simétrica o asimétrica, si la mediana se sitúa en el centro de la caja entonces la distribución es simétrica y tanto la media, mediana y moda coinciden.
Precio
diamonds %>%
ggplot( aes( x = price)) +
geom_histogram(
aes(y = ..density..),
color= "Blue", fill= "White", bins = 30
) +
stat_density(geom = "line", colour = "black", size = 1)+
scale_x_continuous(labels = scales::dollar_format()) +
scale_y_continuous(labels = scales::comma_format()) +
stat_density(geom = "line", colour = "black", size = 1) +
ggtitle("Distribución de precio")
diamonds %>%
ggplot( aes( x = price)) +
geom_boxplot(binwidth = 1000, color= "Blue", fill= "White") +
scale_x_continuous(labels = scales::dollar_format()) +
scale_y_continuous(labels = scales::comma_format()) +
ggtitle("Distribución de precio")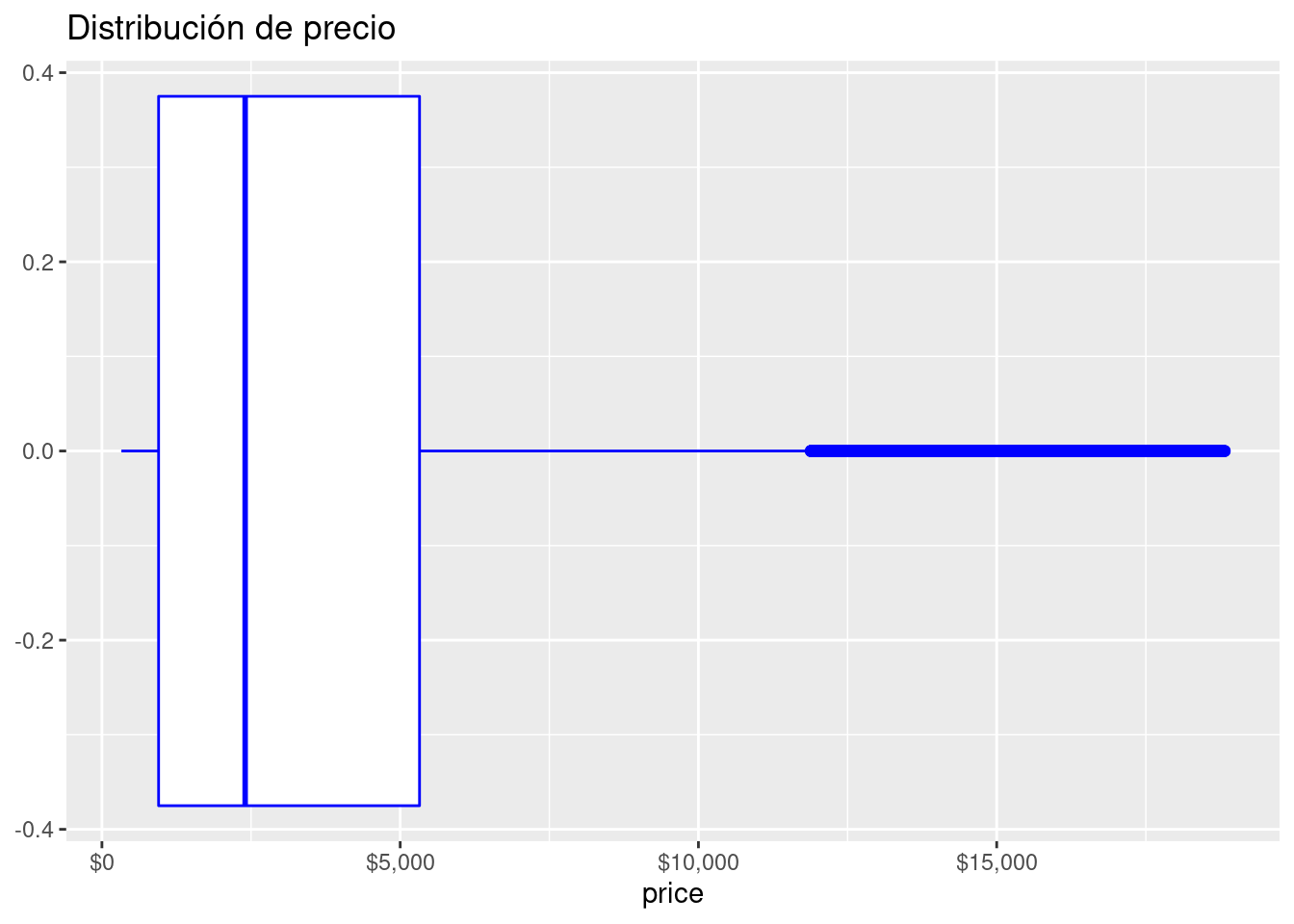
Peso del diamante
diamonds %>%
ggplot( aes( x = carat)) +
geom_histogram(binwidth = .03, color= "purple", fill= "pink", alpha= 0.3) +
scale_y_continuous(labels = scales::comma_format()) +
ggtitle("Distribución de peso de los diamantes") +
theme_bw()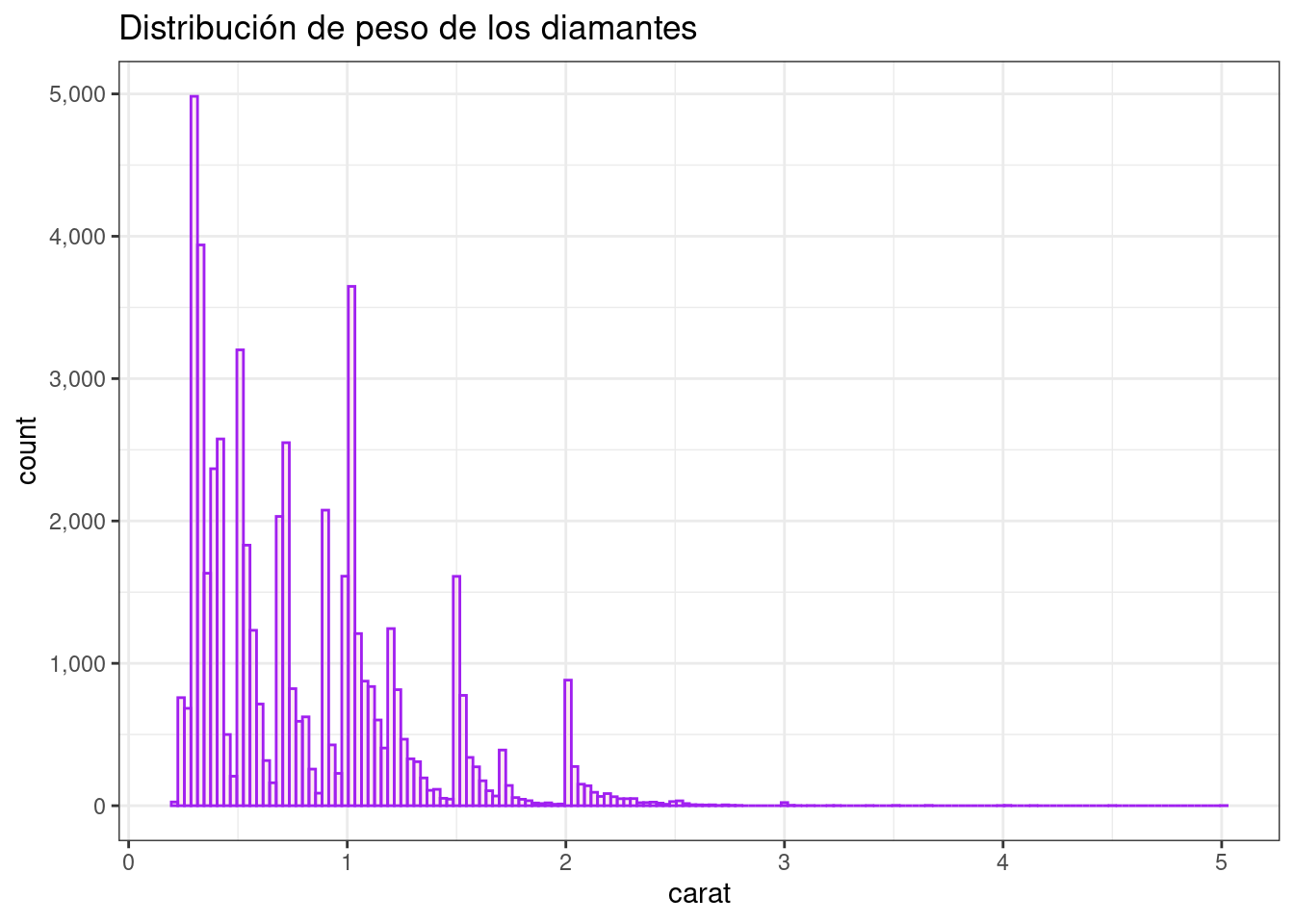
diamonds %>%
ggplot( aes( x = carat)) +
geom_boxplot(binwidth = .3, color= "purple", fill= "pink", alpha= 0.3) +
scale_x_continuous(labels = scales::comma_format()) +
ggtitle("Distribución de peso de los diamantes") +
theme_bw()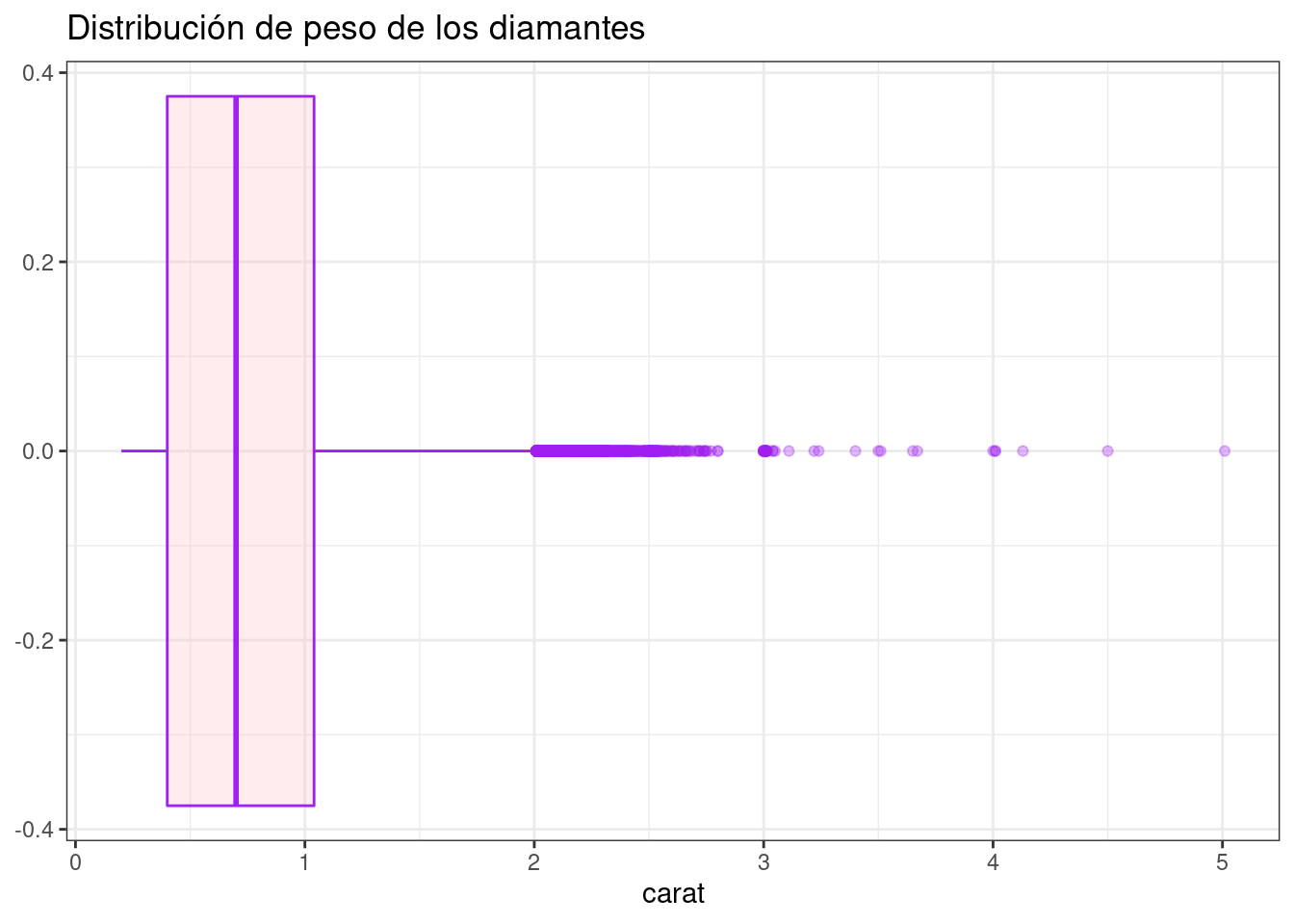
4.1.5.4 Variables nominales/categóricas
Cálidad de corte
diamonds %>%
ggplot( aes( x = cut)) +
geom_bar( color= "darkblue", fill= "cyan", alpha= 0.7) +
scale_y_continuous(labels = scales::comma_format()) +
ggtitle("Distribución de cálidad de corte") +
theme_dark()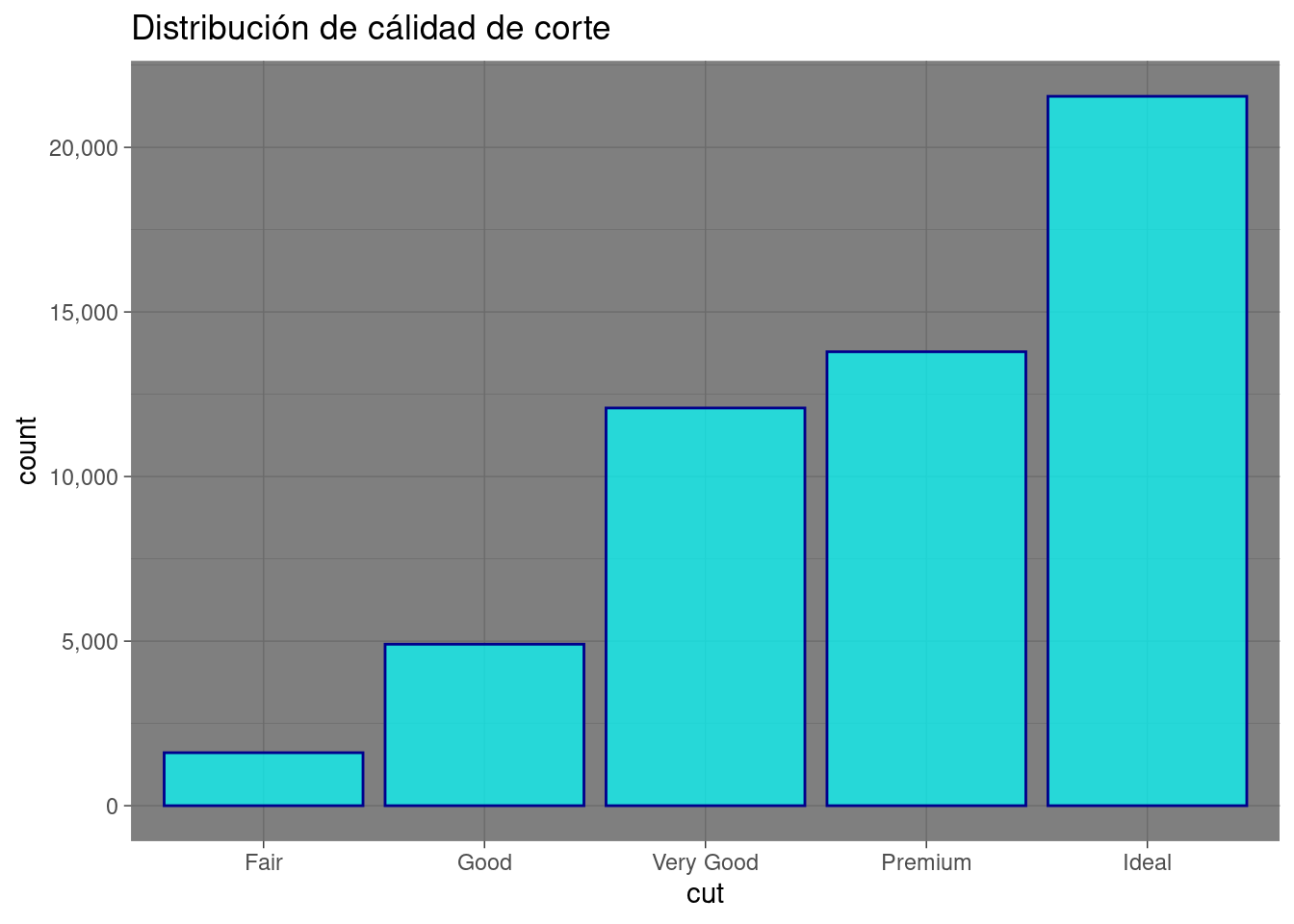
df_pie <- diamonds %>%
group_by(cut) %>%
summarise(freq = n(), .groups='drop')
df_pie %>%
ggplot( aes( x = "", y=freq, fill = factor(cut))) +
geom_bar(width = 1, stat = "identity") +
coord_polar(theta = "y", start=0)
ggplot(data = diamonds)+
geom_bar( mapping = aes(x = cut, fill = cut), show.legend = F, width = 1)+
theme(aspect.ratio = 1)+
labs(x= NULL, y = NULL)+
coord_polar()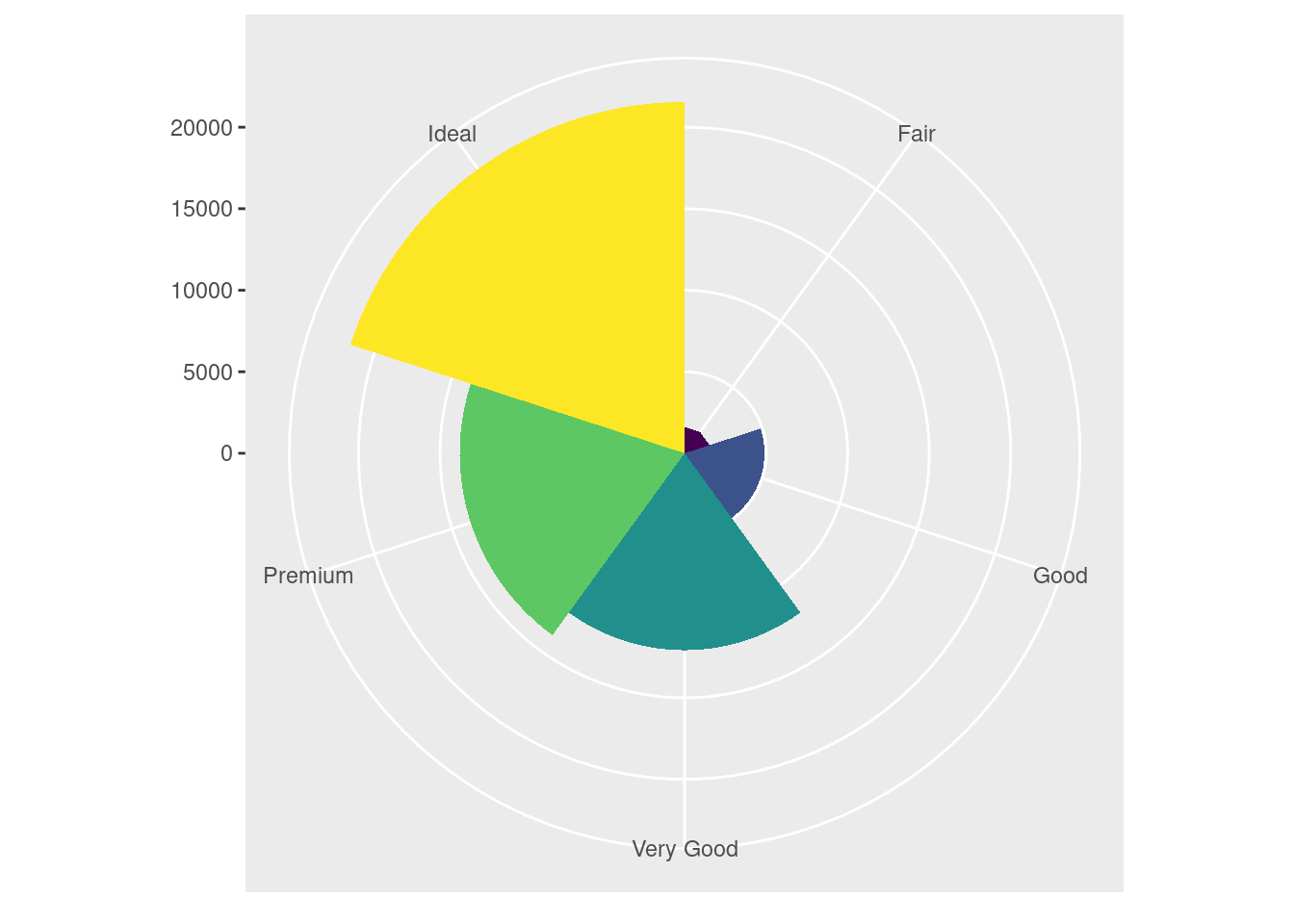
Claridad
diamonds %>%
ggplot( aes( y = clarity)) +
geom_bar( color= "darkblue", fill= "black", alpha= 0.7) +
geom_text(aes(label = ..count..), stat = "count", vjust = 1, hjust = 1.5,colour = "blue")+
scale_x_continuous(labels = scales::comma_format()) +
ggtitle("Distribución claridad") +
theme_get()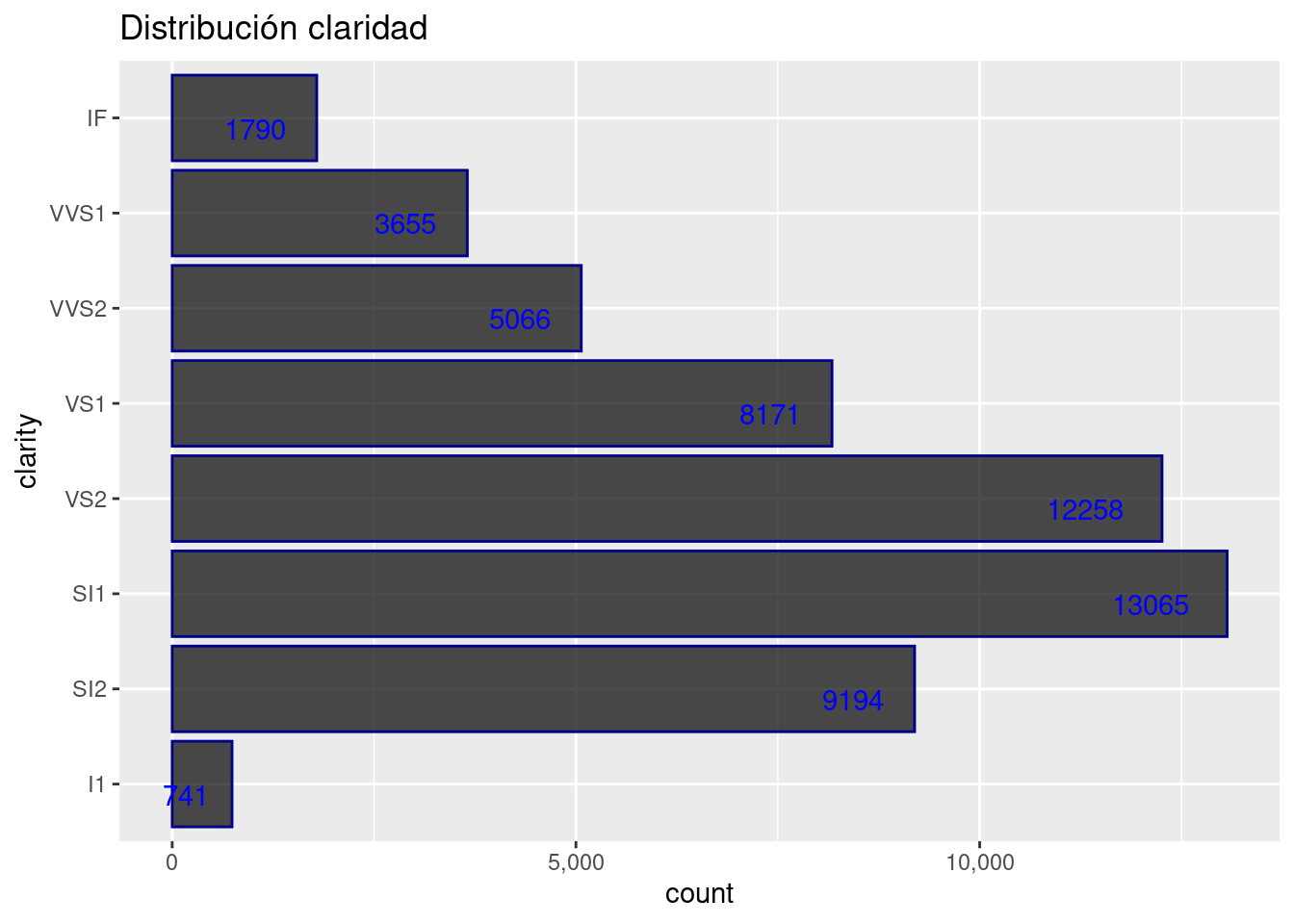
4.1.5.5 Análisis multivariado
Precio vs Calidad del corte
diamonds %>%
ggplot(aes(y= price,x=cut,color=cut)) +
geom_jitter(size=1.2, alpha= 0.5)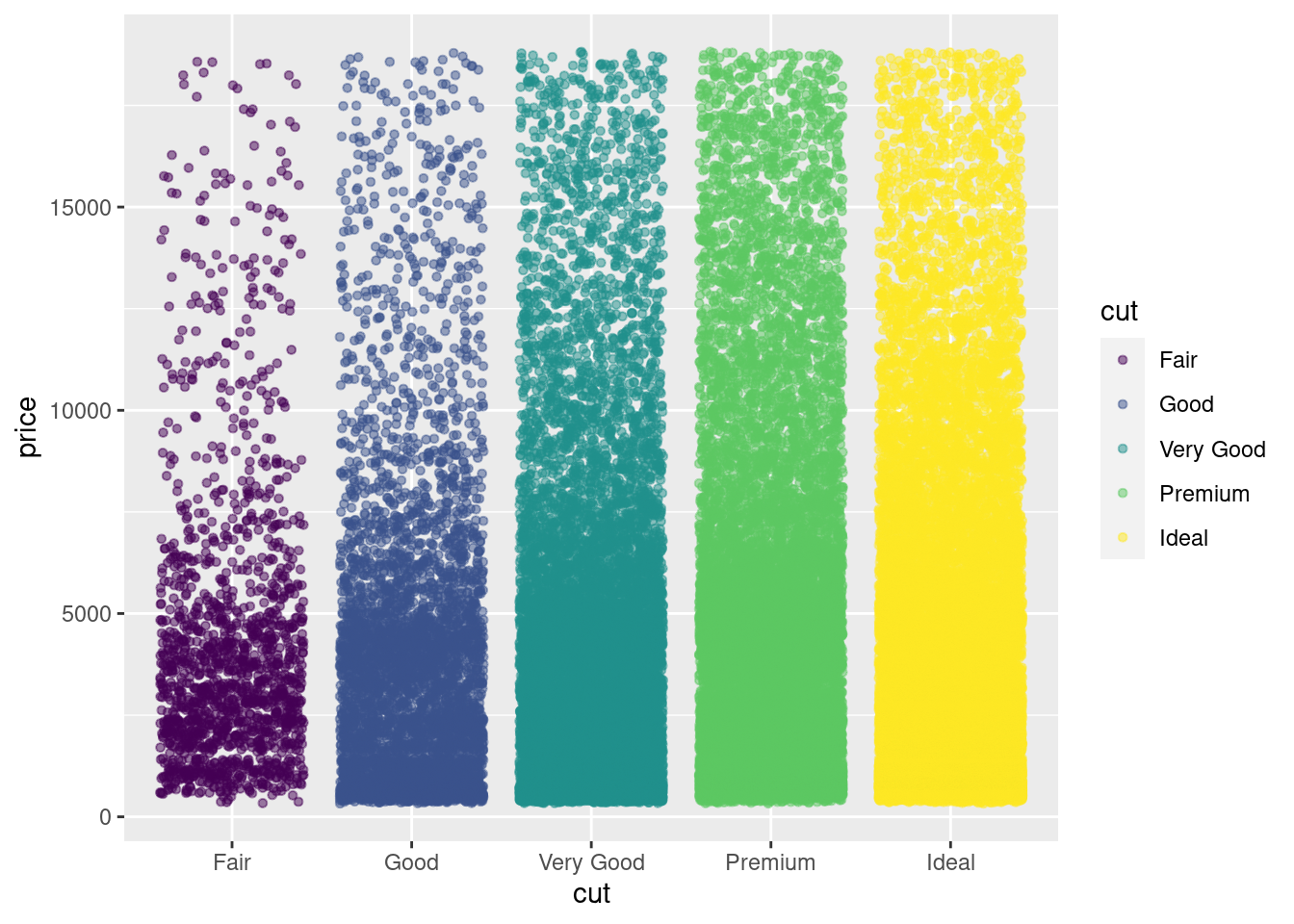
diamonds %>%
ggplot(aes(y= price,x=cut,color=cut)) +
geom_boxplot(size=1.2, alpha= 0.5)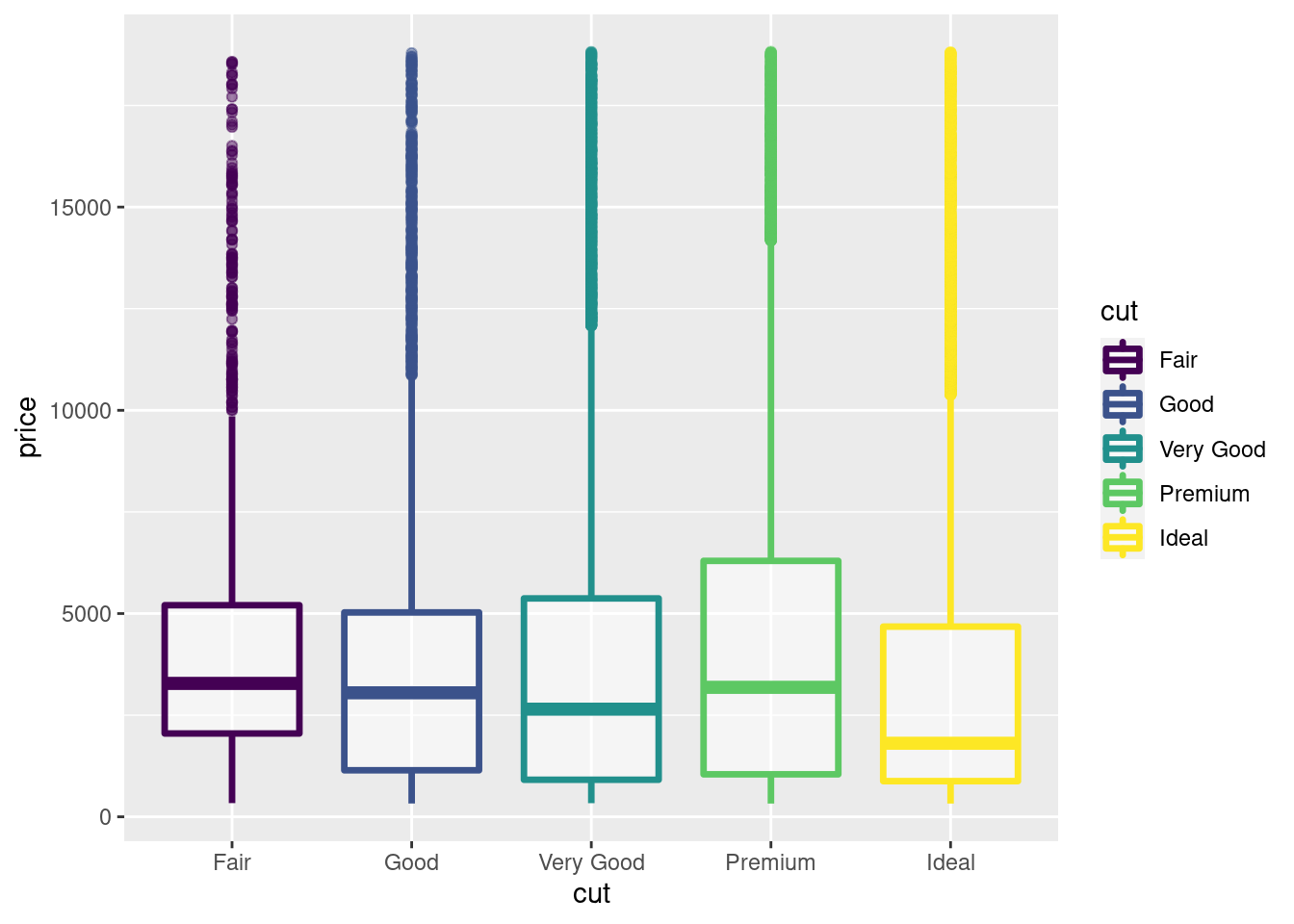
diamonds %>%
ggplot(aes(x= price ,fill=cut)) +
geom_histogram(position = 'identity',
alpha = 0.5)
diamonds %>%
ggplot(aes(x= price ,fill=cut)) +
geom_histogram(position = 'identity',
alpha = 0.5)+
facet_wrap(~cut, ncol = 1)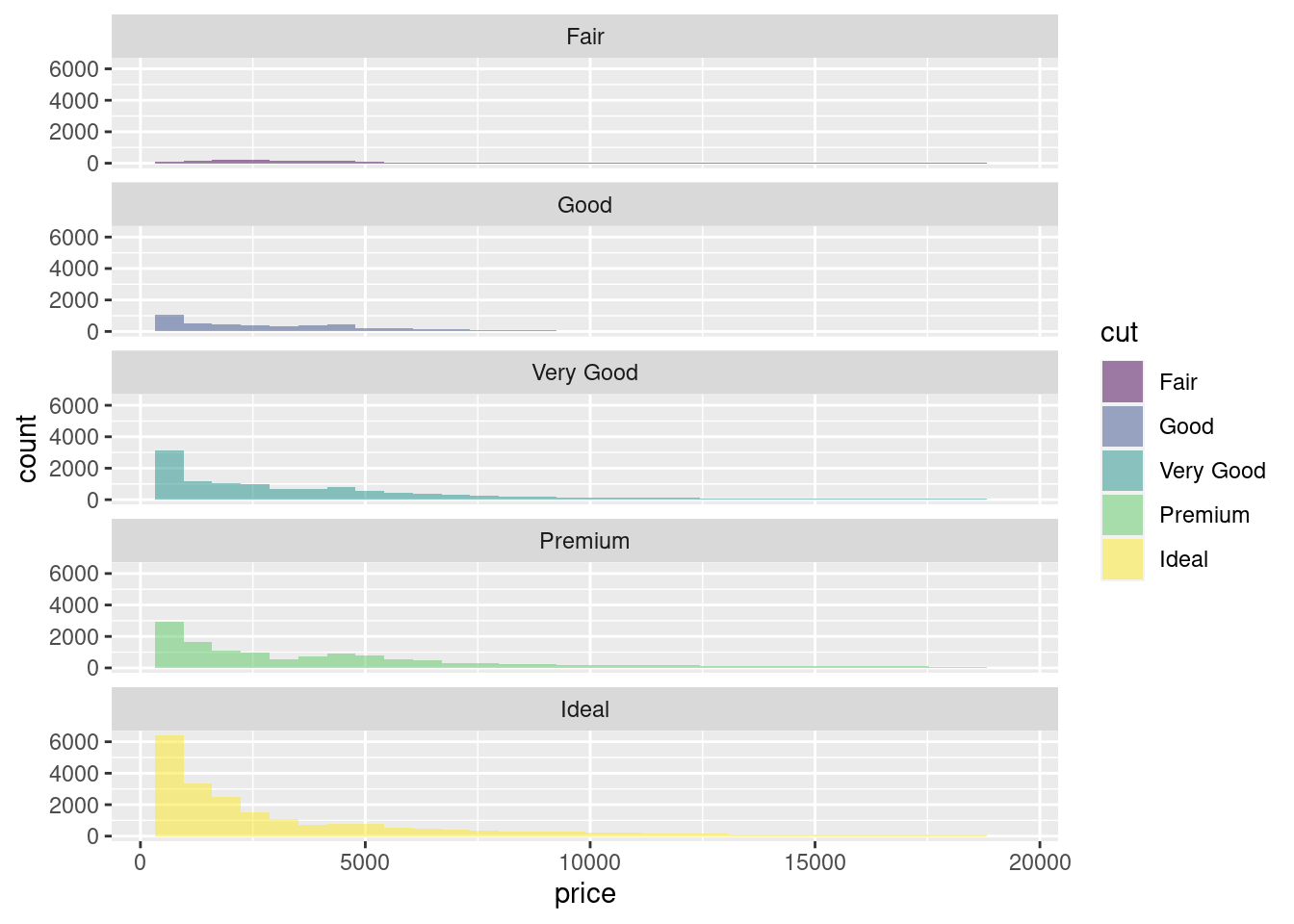
diamonds %>%
ggplot( aes(x = carat ,y=price)) +
geom_point(aes(col = clarity) ) +
geom_smooth()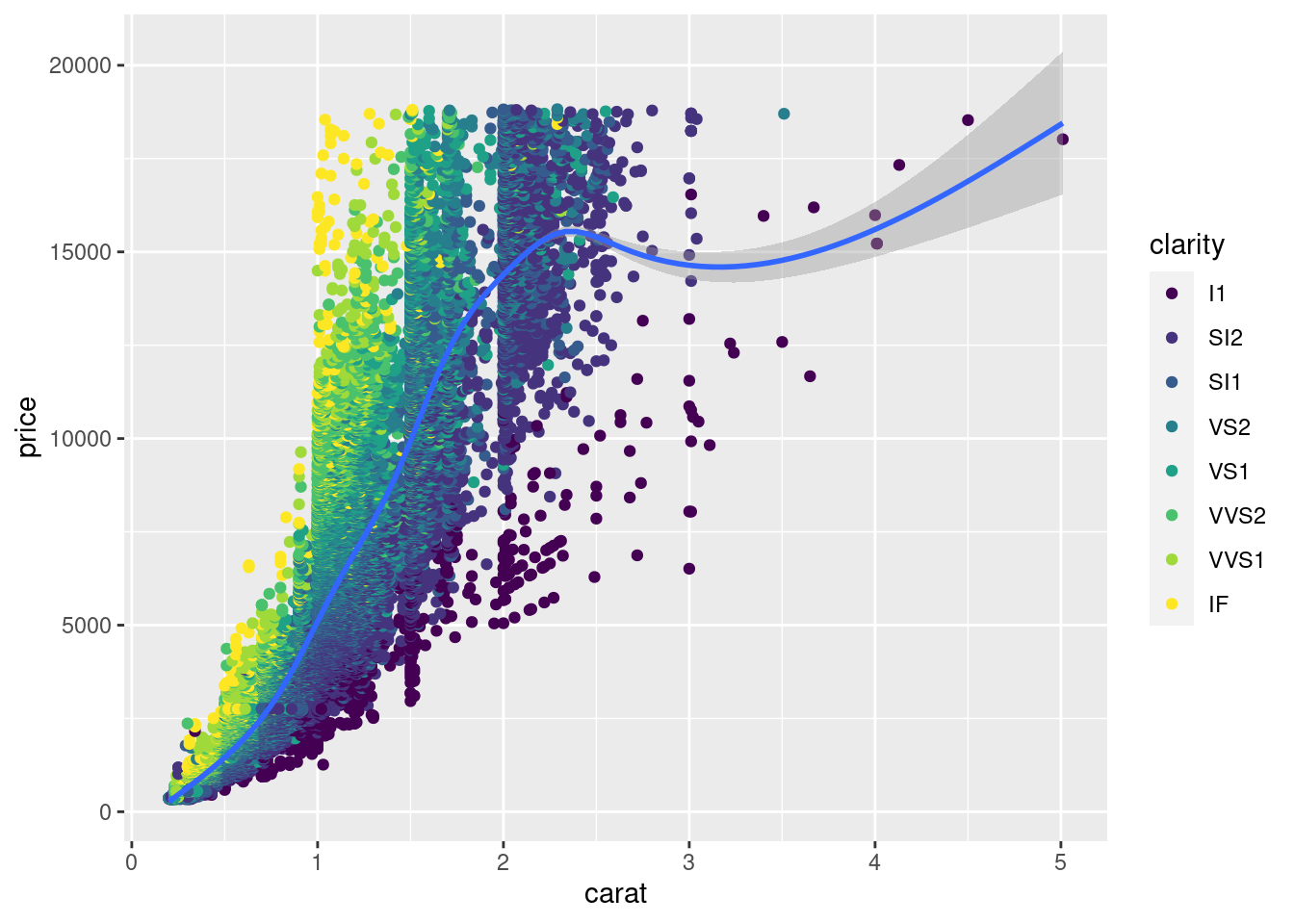
diamonds %>%
ggplot( aes(x = carat ,y=price)) +
geom_point(aes(col = clarity) ) +
facet_wrap(~clarity)+
geom_smooth()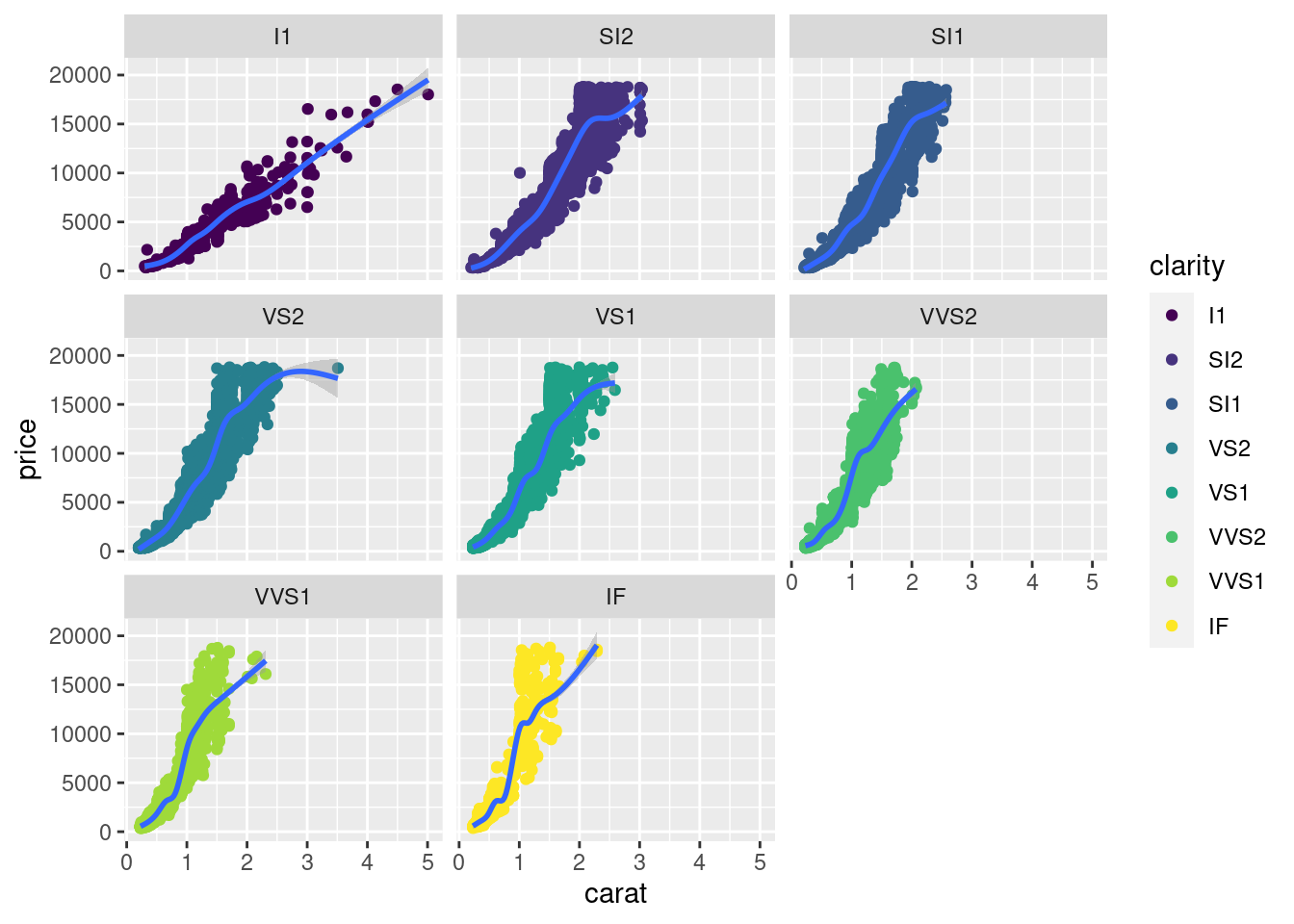
Otro tipo de gráficas interactivas
library(stringr)
fun_mean <- function(x){
mean <- data.frame(
y = mean(x),
label = mean(x, na.rm = T)
)
return(mean)
}
means <- diamonds %>%
group_by(clarity) %>%
summarise(price = round(mean(price), 1))
plot <- diamonds %>%
ggplot(aes(x = clarity, y = price)) +
geom_boxplot(aes(fill = clarity)) +
stat_summary(
fun = mean,
geom = "point",
colour = "darkred",
shape = 18,
size = 3
) +
geom_text(
data = means,
aes(label = str_c("$",price), y = price + 600)
) +
ggtitle("Precio vs Claridad de diamantes") +
xlab("Claridad") +
ylab("Precio")
plotly::ggplotly(plot)4.1.5.6 Warning !
Nunca se debe olvidar que debemos de analizar los datos de manera objetiva, nuestro criterio sobre un problema o negocio no debe de tener sesgos sobre lo que “nos gustaria encontrar en los datos” o lo “que creemos que debe pasar”….
4.2 ML y Algoritmos
Como se habia mencionado, el Machine Learning es una disciplina del campo de la Inteligencia Artificial que, a través de algoritmos, dota a los ordenadores de la capacidad de identificar patrones en datos para hacer predicciones. Este aprendizaje permite a los computadores realizar tareas específicas de forma autónoma.
El término se utilizó por primera vez en 1959. Sin embargo, ha ganado relevancia en los últimos años debido al aumento de la capacidad de computación y al BOOM de los datos.
Un algoritmo para computadoras puede ser pensado como una receta. Describe exactamente qué pasos se realizan uno tras otro. Los ordenadores no entienden las recetas de cocina, sino los lenguajes de programación: En ellos, el algoritmo se descompone en pasos formales (comandos) que el ordenador puede entender.

Algunos problemas pueden formularse fácilmente como un algoritmo, por ejemplo, contando del 1 al 100 o comprobando si un número es un número primo. Para otros problemas, esto es muy difícil, por ejemplo, reconocer la escritura o el texto de las teclas. Aquí los procedimientos de aprendizaje de la máquina ayudan. Durante mucho tiempo se han desarrollado algoritmos que permiten analizar los datos existentes y aplicar los conocimientos derivados de ello a los nuevos datos.
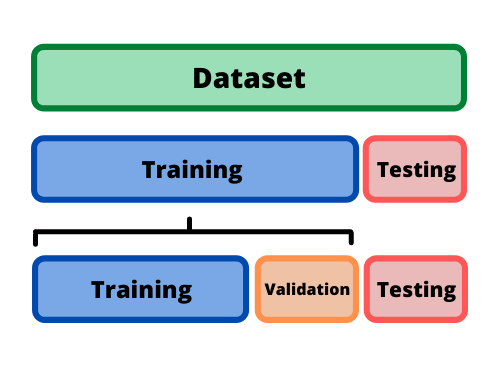
La cuestión no es solo saber para qué sirve el Machine Learning, sino que saber cómo funciona y cómo poder implementarlo en la industria para aprovecharse de sus beneficios. Hay ciertos pasos que usualmente se siguen para crear un modelo de Machine Learning. Estos son típicamente realizados por científicos de los datos que trabajan en estrecha colaboración con los profesionales de los negocios para los que se está desarrollando el modelo.
- Seleccionar y preparar un conjunto de datos de entrenamiento
Los datos de entrenamiento son un conjunto de datos representativos de los datos que el modelo de Machine Learning ingerirá para resolver el problema que está diseñado para resolver.
Los datos de entrenamiento deben prepararse adecuadamente: aleatorizados y comprobados en busca de desequilibrios o sesgos que puedan afectar al entrenamiento. También deben dividirse en dos subconjuntos: el subconjunto de entrenamiento, que se utilizará para entrenar el algoritmo, y el subconjunto de validación, que se utilizará para probarlo y perfeccionarlo.
- Elegir un algoritmo para ejecutarlo en el conjunto de datos de entrenamiento
Este es uno de los pasos más importantes, ya que se debe elegir qué algoritmo utilizar, siendo este un conjunto de pasos de procesamiento estadístico. El tipo de algoritmo depende del tipo (supervisado o no supervisado), la cantidad de datos del conjunto de datos de entrenamiento y del tipo de problema que se debe resolver.


- Entrenamiento del algoritmo para crear el modelo
El entrenamiento del algoritmo es un proceso iterativo: implica ejecutar las variables a través del algoritmo, comparar el resultado con los resultados que debería haber producido, ajustar los pesos y los sesgos dentro del algoritmo que podrían dar un resultado más exacto, y ejecutar las variables de nuevo hasta que el algoritmo devuelva el resultado correcto la mayoría de las veces. El algoritmo resultante, entrenado y preciso, es el modelo de Machine Learning.

- Usar y mejorar el modelo
El paso final es utilizar el modelo con nuevos datos y, en el mejor de los casos, para que mejore en precisión y eficacia con el tiempo. De dónde procedan los nuevos datos dependerá del problema que se resuelva. Por ejemplo, un modelo de Machine Learning diseñado para identificar el spam ingerirá mensajes de correo electrónico, mientras que un modelo de Machine Learning que maneja una aspiradora robot ingerirá datos que resulten de la interacción en el mundo real con muebles movidos o nuevos objetos en la habitación.

4.3 Análisis Supervisado vs No supervisado
Los algoritmos de Machine Learning se dividen en tres categorías, siendo las dos primeras las más comunes:
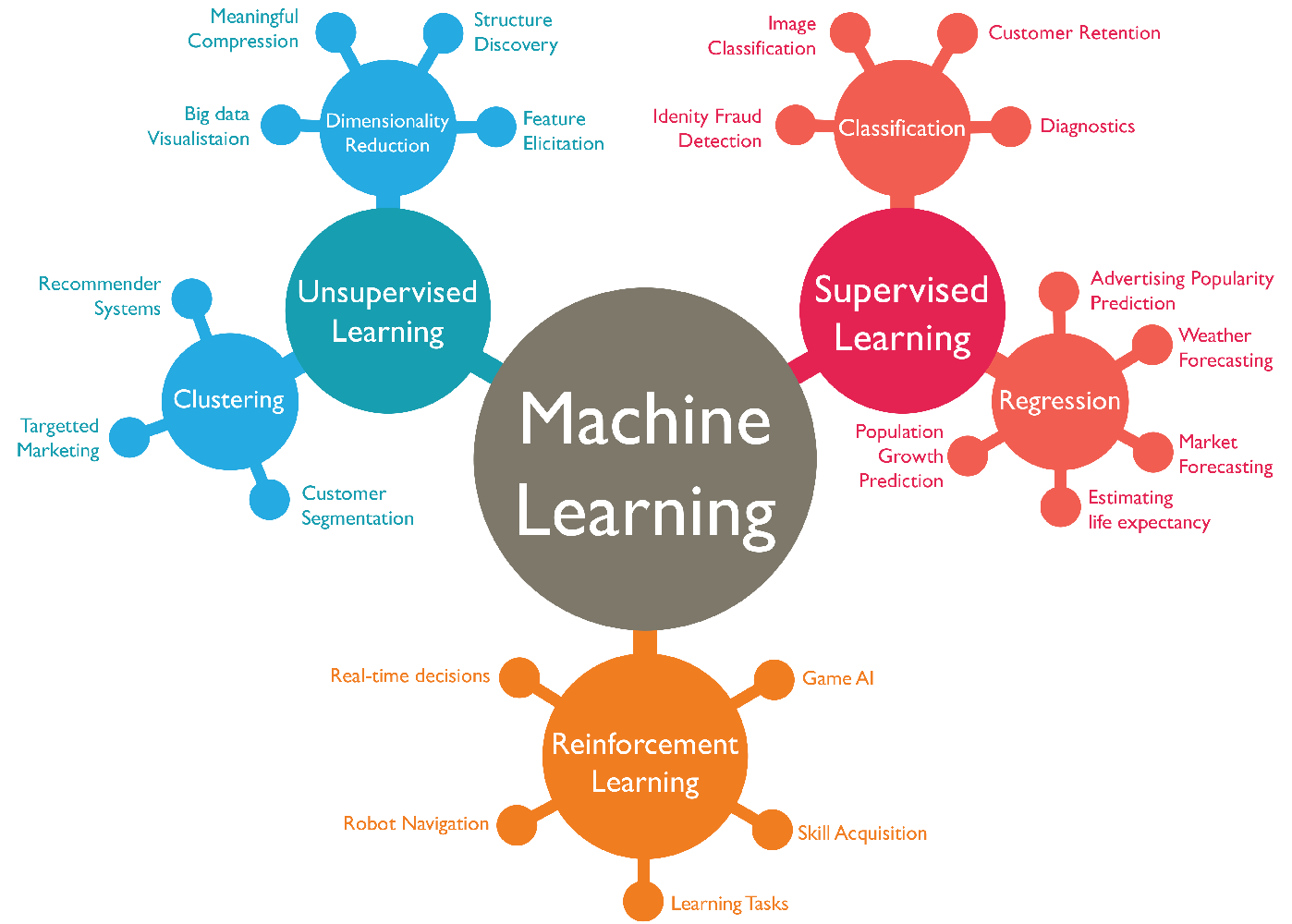
- Aprendizaje supervisado: estos algoritmos cuentan con un aprendizaje previo basado en un sistema de etiquetas asociadas a unos datos que les permiten tomar decisiones o hacer predicciones.
Algunos ejemplos son:
- Un detector de spam que etiqueta un e-mail como spam o no.
- Predecir precios de casas
- Clasificación de imagenes
- Predecir el clima
- ¿Quiénes son los clientes descontentos?- Aprendizaje no supervisado: en el aprendizaje supervisado, la idea principal es aprender bajo supervisión, donde la señal de supervisión se nombra como valor objetivo o etiqueta. En el aprendizaje no supervisado, carecemos de este tipo de etiqueta. Por lo tanto, necesitamos encontrar nuestro camino sin ninguna supervisión ni guía. Esto simplemente significa que necesitamos descubrir qué es qué por nosotros mismos.
Algunos ejemplos son:
- Encontrar segmentos de clientes.
- Reducir la complejidad de un problema
- Selección de variables
- Encontrar grupos
- Reducción de dimensionalidad- Aprendizaje por refuerzo: su objetivo es que un algoritmo aprenda a partir de la propia experiencia. Esto es, que sea capaz de tomar la mejor decisión ante diferentes situaciones de acuerdo a un proceso de prueba y error en el que se recompensan las decisiones correctas.
Algunos ejemplos son:
- Reconocimiento facial
- Diagnósticos médicos
- Clasificar secuencias de ADN4.3.1 Regresión vs clasificación
Existen dos tipos principales de aprendizaje supervisado, esto depende del tipo de la variable respuesta:
4.3.1.1 Clasificación
En el aprendizaje supervisado, los algoritmos de clasificación se usan cuando el resultado es una etiqueta discreta. Esto quiere decir que se utilizan cuando la respuesta se fundamenta en conjunto finito de resultados.
4.3.1.2 Regresión
El análisis de regresión es un subcampo del aprendizaje automático supervisado cuyo objetivo es establecer un método para la relación entre un cierto número de características y una variable objetivo continua.
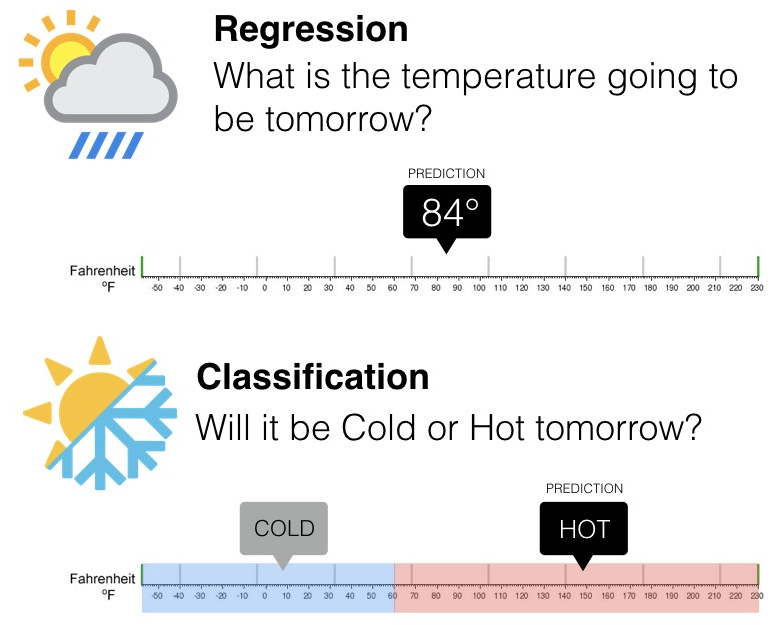
4.4 Sesgo vs varianza
En el mundo de Machine Learning cuando desarrollamos un modelo nos esforzamos para hacer que sea lo más preciso, ajustando los parámetros, pero la realidad es que no se puede construir un modelo 100% preciso ya que nunca pueden estar libres de errores.
Comprender cómo las diferentes fuentes de error generan sesgo y varianza nos ayudará a mejorar el proceso de ajuste de datos, lo que resulta en modelos más precisos, adicionalmente también evitará el error de sobreajuste y falta de ajuste.
4.4.0.1 Errores reducibles
- Error por sesgo:
Es la diferencia entre la predicción esperada de nuestro modelo y los valores verdaderos. Aunque al final nuestro objetivo es siempre construir modelos que puedan predecir datos muy cercanos a los valores verdaderos, no siempre es tan fácil porque algunos algoritmos son simplemente demasiado rígidos para aprender señales complejas del conjunto de datos.
Imagina ajustar una regresión lineal a un conjunto de datos que tiene un patrón no lineal, no importa cuántas observaciones más recopiles, una regresión lineal no podrá modelar las curvas en esos datos. Esto se conoce como underfitting.
- Error por varianza:
Se refiere a la cantidad que la estimación de la función objetivo cambiará si se utiliza diferentes datos de entrenamiento. La función objetivo se estima a partir de los datos de entrenamiento mediante un algoritmo de Machine Learning, por lo que deberíamos esperar que el algoritmo tenga alguna variación. Idealmente no debería cambiar demasiado de un conjunto de datos de entrenamiento a otro.
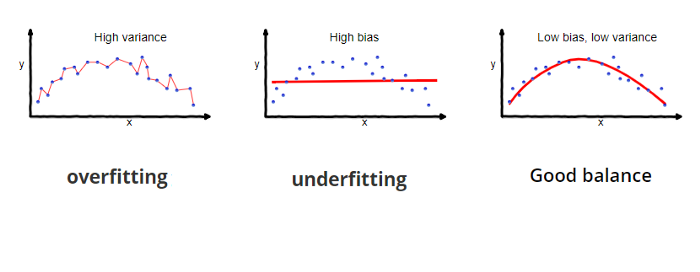
Los algoritmos de Machine Learning que tienen una gran varianza están fuertemente influenciados por los detalles de los datos de entrenamiento, esto significa que los detalles de la capacitación influyen en el número y los tipos de parámetros utilizados para caracterizar la función de mapeo.
4.4.0.2 Error irreducible
El error irreducible no se puede reducir, independientemente de qué algoritmo se usa. También se le conoce como ruido y, por lo general, proviene por factores como variables desconocidas que influyen en el mapeo de las variables de entrada a la variable de salida, un conjunto de características incompleto o un problema mal enmarcado. Acá es importante comprender que no importa cuán bueno hagamos nuestro modelo, nuestros datos tendrán cierta cantidad de ruido o un error irreductible que no se puede eliminar.
4.4.0.3 Balance entre sesgo y varianza o Trade-off
El objetivo de cualquier algoritmo supervisado de Machine Learning es lograr un bias bajo, una baja varianza y a su vez el algoritmo debe lograr un buen rendimiento de predicción.
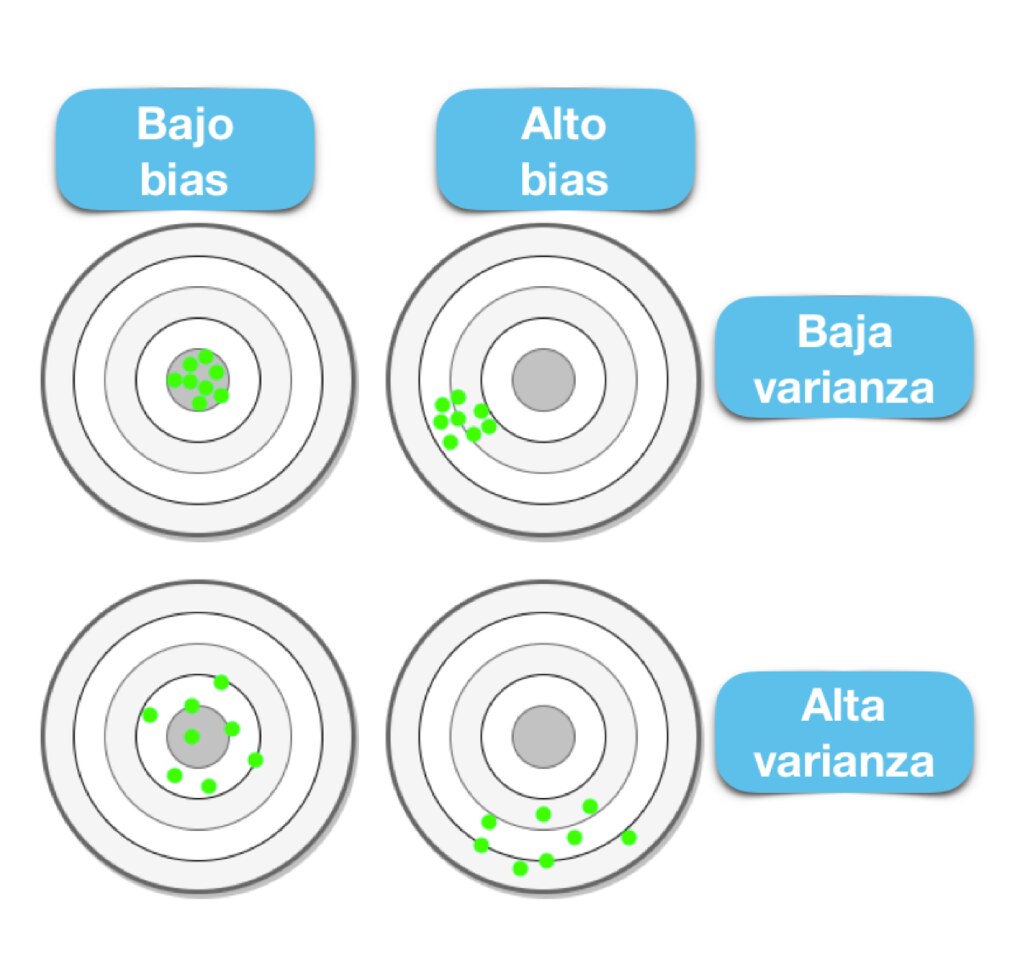
El bias frente a la varianza se refiere a la precisión frente a la consistencia de los modelos entrenados por su algoritmo. Podemos diagnosticarlos de la siguiente manera:

Los algoritmos de baja varianza (alto bias) tienden a ser menos complejos, con una estructura subyacente simple o rígida.
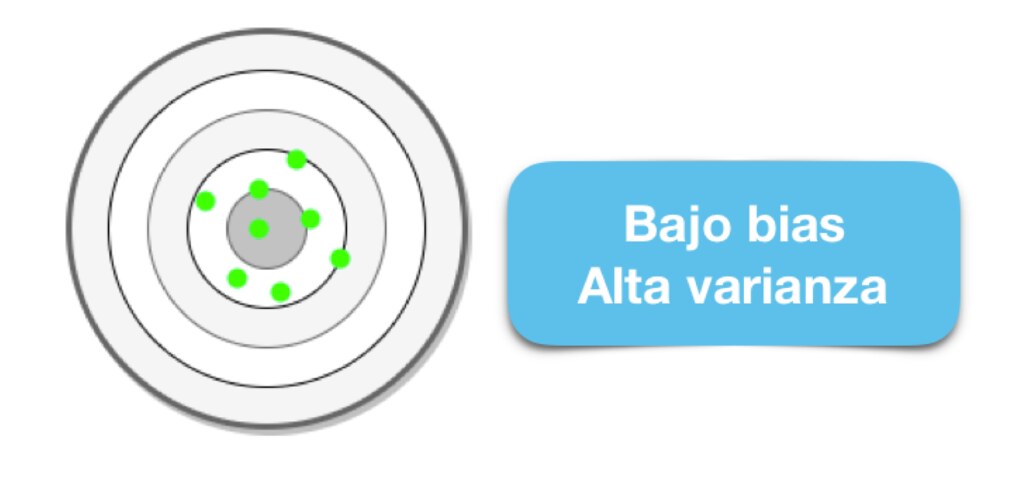
Los algoritmos de bajo bias (alta varianza) tienden a ser más complejos, con una estructura subyacente flexible.
No hay escapatoria a la relación entre el bias y la varianza en Machine Learning, aumentar el bias disminuirá la varianza, aumentar la varianza disminuirá el bias.
4.4.0.4 Error total
Comprender el sesgo y la varianza es fundamental para comprender el comportamiento de los modelos de predicción, pero en general lo que realmente importa es el error general, no la descomposición específica. El punto ideal para cualquier modelo es el nivel de complejidad en el que el aumento en el sesgo es equivalente a la reducción en la varianza.
Para construir un buen modelo, necesitamos encontrar un buen equilibrio entre el bias y la varianza de manera que minimice el error total.
Un equilibrio óptimo de bias y varianza nunca sobreequiparía o no sería adecuado para el modelo. Por lo tanto comprender el bias y la varianza es fundamental para comprender el comportamiento de los modelos de predicción.

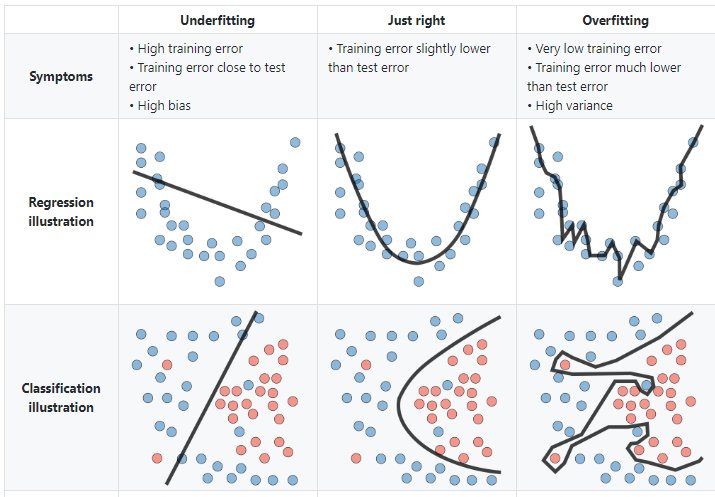
4.4.0.5 Overfitting
El modelo es muy particular.
Error debido a la varianza
Durante el entrenamiento tiene un desempeño muy bueno, pero al pasar nuevos datos su desempeño es malo.
4.4.0.6 Underfitting
El modelo es demasiado general.
Error debido al sesgo.
Durante el entrenamiento no tiene un buen desempeño.
4.5 Pre-procesamiento e ingeniería de datos
Hay varios pasos que se deben de seguir para crear un modelo útil:
- Recopilación de datos.
- Limpieza de datos.
- Creación de nuevas variables.
- Estimación de parámetros.
- Selección y ajuste del modelo.
- Evaluación del rendimiento.
Al comienzo de un proyecto, generalmente hay un conjunto finito de datos disponibles para todas estas tareas.
OJO: A medida que los datos se reutilizan para múltiples tareas, aumentan los riesgos de agregar sesgos o grandes efectos de errores metodológicos.
4.5.1 Pre-procesamiento de datos
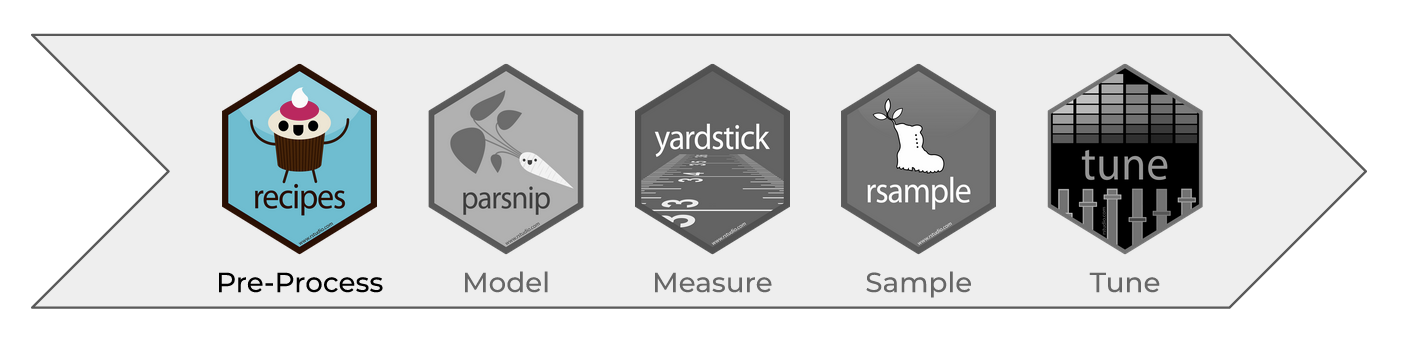
Como punto de partida para nuestro flujo de trabajo de aprendizaje automático, necesitaremos datos de entrada. En la mayoría de los casos, estos datos se cargarán y almacenarán en forma de data frames o tibbles en R. Incluirán una o varias variables predictoras y, en caso de aprendizaje supervisado, también incluirán un resultado conocido.
Sin embargo, no todos los modelos pueden lidiar con diferentes problemas de datos y, a menudo, necesitamos transformar los datos para obtener el mejor rendimiento posible del modelo. Este proceso se denomina pre-procesamiento y puede incluir una amplia gama de pasos, como:
- Dicotomización de variables: Variables cualitativas que solo pueden tomar el valor \(0\) o \(1\) para indicar la ausencia o presencia de una condición específica. Estas variables se utilizan para clasificar los datos en categorías mutuamente excluyentes o para activar comandos de encendido / apagado


- Near Zero Value (nzv) o Varianza Cero: En algunas situaciones, el mecanismo de generación de datos puede crear predictores que solo tienen un valor único (es decir, un “predictor de varianza cercando a cero”). Para muchos modelos (excluidos los modelos basados en árboles), esto puede hacer que el modelo se bloquee o que el ajuste sea inestable.
De manera similar, los predictores pueden tener solo una pequeña cantidad de valores únicos que ocurren con frecuencias muy bajas.

- Imputaciones: Si faltan algunos predictores, ¿deberían estimarse mediante imputación?
 * Des-correlacionar: Si hay predictores correlacionados, ¿debería mitigarse esta correlación? Esto podría significar filtrar predictores, usar análisis de componentes principales o una técnica basada en modelos (por ejemplo, regularización).
* Des-correlacionar: Si hay predictores correlacionados, ¿debería mitigarse esta correlación? Esto podría significar filtrar predictores, usar análisis de componentes principales o una técnica basada en modelos (por ejemplo, regularización).
 * Normalizar: ¿Deben centrarse y escalar los predictores?
* Normalizar: ¿Deben centrarse y escalar los predictores?
- Transformar: ¿Es útil transformar los predictores para que sean más simétricos? (por ejemplo, escala logarítmica).
Dependiendo del caso de uso, algunos pasos de pre-procesamiento pueden ser indispensables para pasos posteriores, mientras que otros solo son opcionales. Sin embargo, dependiendo de los pasos de pre-procesamiento elegidos, el rendimiento del modelo puede cambiar significativamente en pasos posteriores. Por lo tanto, es muy común probar varias configuraciones.
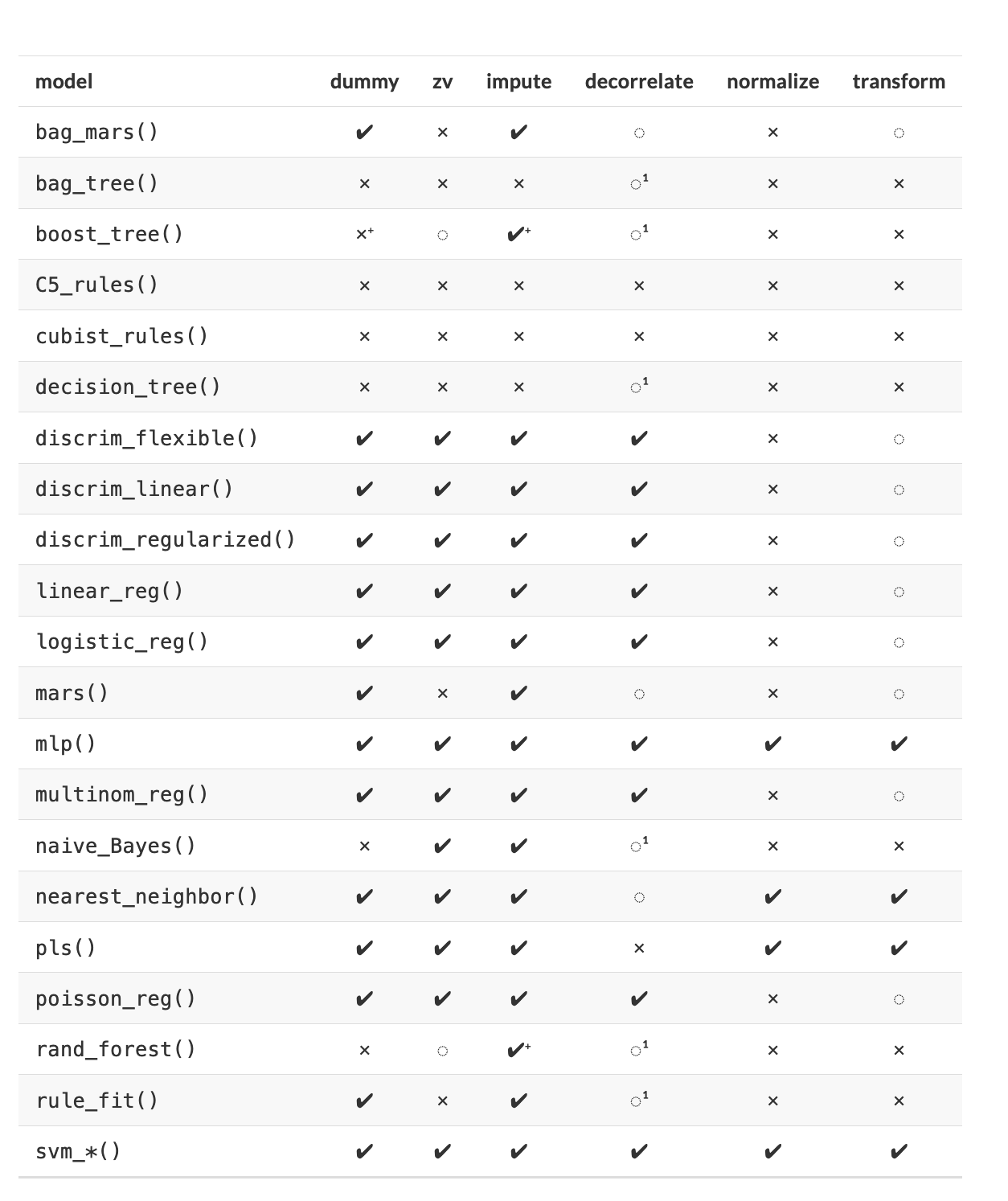
En la tabla, \(\checkmark\) indica que el método es obligatorio para el modelo y \(\times\) indica que no lo es. El símbolo \(\circ\) significa que la técnica puede ayudar al modelo, pero no es obligatorio.
 Notación:
Notación:
- Es posible que la des-correlación de predictores no ayude a mejorar el rendimiento. Sin embargo, menos predictores correlacionados pueden mejorar la estimación de las puntuaciones de importancia de la varianza. Esencialmente, la selección de predictores altamente correlacionados es casi aleatoria.
La notación \(+\) significa que la respuesta depende de la implementación:
Teoricamente, cualquier modelo basado en árboles no requiere imputación de datos, sin embargo, muchas implementaciones de conjuntos de árboles requieren imputación.
Si bien los métodos de refuerzo basados en árboles generalmente no requieren la creación de variables ficticias, los modelos que usan
xgboostsí lo hacen.
4.5.2 Ingeniería de datos
La ingeniería de datos abarca actividades que reformatean los valores de los predictores para que se puedan utilizar de manera eficaz para nuestro modelo. Esto incluye transformaciones y codificaciones de los datos para representar mejor sus características importantes.
Por ejemplo:
1.- Supongamos que un conjunto de datos tiene dos predictores que se pueden representar de manera más eficaz en nuestro modelo como una proporción, así, tendríamos un nuevo predictor a partir de la proporción de los dos predictores originales.
| x | x_prop |
|---|---|
| 691 | 0.1836789 |
| 639 | 0.1698565 |
| 969 | 0.2575758 |
| 955 | 0.2538543 |
| 508 | 0.1350346 |
2.- Al elegir cómo codificar nuestros datos en el modelado, podríamos elegir una opción que creemos que está más asociada con el resultado. El formato original de los datos, por ejemplo numérico (edad) versus categórico (grupo).
| Edad | Grupo |
|---|---|
| 7 | Niños |
| 78 | Adultos mayores |
| 17 | Adolescentes |
| 25 | Adultos |
| 90 | Adultos mayores |
La ingeniería y el pre-procesamiento de datos también pueden implicar el reformateo requerido por el modelo. Algunos modelos utilizan métricas de distancia geométrica y, en consecuencia, los predictores numéricos deben centrarse y escalar para que estén todos en las mismas unidades. De lo contrario, los valores de distancia estarían sesgados por la escala de cada columna.
4.6 Recetas

Una receta es una serie de pasos o instrucciones para el procesamiento de datos. A diferencia del método de fórmula dentro de una función de modelado, la receta define los pasos sin ejecutarlos inmediatamente; es sólo una especificación de lo que se debe hacer. La estructura de una receta sigue los siguientes pasos:
Inicialización
Transformación
Preparación
Aplicación
La siguiente sección explica la estructura y flujo de transformaciones:
receta <- recipe(response ~ X1 + X2 + X3 + ... + Xn, data = dataset ) %>%
transformation_1(...) %>%
transformation_2(...) %>%
transformation_3(...) %>%
...
final_transformation(...) %>%
prep()
bake(receta, new_data = new_dataset)A continuación se muestran distintos ejemplos de transformaciones realizadas comunmente en el pre-procesamiento de modelos predictivos. Como ejemplo, utilizaremos el subconjunto de predictores disponibles en los datos de vivienda: Ames
Vecindario (29 vecindarios)
Superficie habitable bruta sobre el nivel del suelo
Año de constricción
Tipo de edificio
ANTERIORMENTE… Un modelo de regresión lineal ordinario se ajustaba a los datos con la función estándar lm() de la siguiente manera:
lm(Sale_Price ~ Neighborhood + log10(Gr_Liv_Area) + Year_Built + Bldg_Type, data = ames)Cuando se ejecuta esta función, los datos se convierten en a una matriz de diseño numérico (también llamada matriz de modelo) y luego se utiliza el método de mínimos cuadrados para estimar los parámetros. Lo que hace la fórmula anterior se puede descomponer en una serie de pasos:
1.- El precio de venta se define como el resultado, mientras que las variables de vecindario, superficie habitable bruta, año de construcción y tipo de edificio se definen como predictores.
2.- Se aplica una transformación logarítmica al predictor de superficie habitable bruta.
3.- Las columnas de vecindad y tipo de edificio se convierten de un formato no numérico a un formato numérico (dado que los mínimos cuadrados requieren predictores numéricos).
La siguiente receta es equivalente a la fórmula anterior:
simple_ames <- recipe(
Sale_Price ~ Neighborhood + Gr_Liv_Area + Year_Built + Bldg_Type,
data = ames) %>%
step_log(Gr_Liv_Area, base = 10) %>%
step_dummy(all_nominal_predictors())
simple_ames## Recipe
##
## Inputs:
##
## role #variables
## outcome 1
## predictor 4
##
## Operations:
##
## Log transformation on Gr_Liv_Area
## Dummy variables from all_nominal_predictors()Ventajas de usar una receta:
Los cálculos se pueden reciclar entre modelos ya que no están estrechamente acoplados a la función de modelado.
Una receta permite un conjunto más amplio de opciones de procesamiento de datos que las que pueden ofrecer las fórmulas.
La sintaxis puede ser muy compacta. Por ejemplo,
all_nominal_predictors()se puede usar para capturar muchas variables para tipos específicos de procesamiento, mientras que una fórmula requeriría que cada una se enumere explícitamente.Todo el procesamiento de datos se puede capturar en un solo objeto en lugar de tener scripts que se repiten o incluso se distribuyen en diferentes archivos.
4.6.1 Pasos y estructura de recetas
Como se mostró anteriormente, existen 4 pasos fundamentales para el procesamiento y transformación de conjuntos de datos. Estos pasos se describen de la siguiente manera:
Receta: Inicializa una receta y define los roles de las variables
Transformaciones: Mutaciones a los renglones y columnas hasta desear el resultado
Preparación: Se realizan las estimaciones estadísticas con los datos
La función prep() estima las cantidades requeridas y las estadísticas necesarias para cualquier paso declarado en la receta.
prep <- prep(simple_ames)
prep## Recipe
##
## Inputs:
##
## role #variables
## outcome 1
## predictor 4
##
## Training data contained 2930 data points and no missing data.
##
## Operations:
##
## Log transformation on Gr_Liv_Area [trained]
## Dummy variables from Neighborhood, Bldg_Type [trained]- Aplicación Se llevan a cabo las transformaciones especificadas en la receta preparada a un conjunto de datos.
Finalmente, la función bake() lleva a cabo la transformación de un conjunto de datos a través de las estimaciones indicadas en una receta y aplica las operaciones a un conjunto de datos para crear una matriz de diseño. La función bake(object, new_data = NULL) devolverá los datos con los que se entrenó la receta.
Nota: La función juice() devolverá los resultados de una receta en la que se hayan aplicado todos los pasos a los datos. Similar a la función bake() con el comando new_data = NULL.
simple_ames %>%
prep() %>%
bake(new_data = NULL) %>%
glimpse()## Rows: 2,930
## Columns: 35
## $ Gr_Liv_Area <dbl> 3.219060, 2.95230…
## $ Year_Built <int> 1960, 1961, 1958,…
## $ Sale_Price <int> 215000, 105000, 1…
## $ Neighborhood_College_Creek <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Old_Town <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Edwards <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Somerset <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Northridge_Heights <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Gilbert <dbl> 0, 0, 0, 0, 1, 1,…
## $ Neighborhood_Sawyer <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Northwest_Ames <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Sawyer_West <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Mitchell <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Brookside <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Crawford <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Iowa_DOT_and_Rail_Road <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Timberland <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Northridge <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Stone_Brook <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_South_and_West_of_Iowa_State_University <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Clear_Creek <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Meadow_Village <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Briardale <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Bloomington_Heights <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Veenker <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Northpark_Villa <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Blueste <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Greens <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Green_Hills <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Landmark <dbl> 0, 0, 0, 0, 0, 0,…
## $ Neighborhood_Hayden_Lake <dbl> 0, 0, 0, 0, 0, 0,…
## $ Bldg_Type_TwoFmCon <dbl> 0, 0, 0, 0, 0, 0,…
## $ Bldg_Type_Duplex <dbl> 0, 0, 0, 0, 0, 0,…
## $ Bldg_Type_Twnhs <dbl> 0, 0, 0, 0, 0, 0,…
## $ Bldg_Type_TwnhsE <dbl> 0, 0, 0, 0, 0, 0,…En cuanto a las transformaciones posibles, existe una gran cantidad de funciones que soportan este proceso. En esta sección se muestran algunas de las transformación más comunes, entre ellas:
- Normalización
- Dicotomización
- Creación de nuevas columnas
- Datos faltantes
- Imputaciones
- Interacciones
- Etc.
4.6.1.1 Normalizar columnas numéricas
Quizá la transformación numérica más usada en todos los modelos es la estandarización o normalización de variables numéricas. Este proceso se realiza para homologar la escala de las variables numéricas, de modo que no predomine una sobre otra debido a la diferencia de magnitudes o escalas. Este proceso se tiene de fondo el siguiente proceso estadístico:
\[Z=\frac{X-\hat{\mu}_x}{\hat{\sigma}_x}\]
Donde:
X = Es una variable o columna numérica
\(\hat{\mu}_x\) = Es la estimación de la media de la variable X
\(\hat{\sigma}_x\) = Es la estimación de la desviación estándar de la variable X
La librería recipes nos permite realizar este proceso ágilmente mediante la función: step_normalize().
ames %>% select(Sale_Price, Neighborhood, Gr_Liv_Area, Year_Built, Bldg_Type) %>%
head(5)## # A tibble: 5 × 5
## Sale_Price Neighborhood Gr_Liv_Area Year_Built Bldg_Type
## <int> <fct> <int> <int> <fct>
## 1 215000 North_Ames 1656 1960 OneFam
## 2 105000 North_Ames 896 1961 OneFam
## 3 172000 North_Ames 1329 1958 OneFam
## 4 244000 North_Ames 2110 1968 OneFam
## 5 189900 Gilbert 1629 1997 OneFamsimple_ames <- recipe(Sale_Price ~ ., data = ames) %>%
step_normalize(all_numeric_predictors())
simple_ames## Recipe
##
## Inputs:
##
## role #variables
## outcome 1
## predictor 73
##
## Operations:
##
## Centering and scaling for all_numeric_predictors()simple_ames %>%
prep() %>%
bake(new_data = NULL) %>%
select(Sale_Price, Neighborhood, Gr_Liv_Area, Year_Built, Bldg_Type) %>%
head(5)## # A tibble: 5 × 5
## Sale_Price Neighborhood Gr_Liv_Area Year_Built Bldg_Type
## <int> <fct> <dbl> <dbl> <fct>
## 1 215000 North_Ames 0.309 -0.375 OneFam
## 2 105000 North_Ames -1.19 -0.342 OneFam
## 3 172000 North_Ames -0.338 -0.442 OneFam
## 4 244000 North_Ames 1.21 -0.111 OneFam
## 5 189900 Gilbert 0.256 0.848 OneFam4.6.1.2 Dicotomización de categorías
Otra transformación necesaria en la mayoría de los modelos predictivos en la creación de las variables dummy. Se mencionó anteriormente que los modelos requieren de una matriz numérica de características explicativas que permita calcular patrones estadísticos para predecir la variable de respuesta. El proceso de dicotomización consiste en crear una variable dicotómica por cada categoría de una columna con valores nominales.
ames %>% select(Sale_Price, Bldg_Type) %>% head(5)## # A tibble: 5 × 2
## Sale_Price Bldg_Type
## <int> <fct>
## 1 215000 OneFam
## 2 105000 OneFam
## 3 172000 OneFam
## 4 244000 OneFam
## 5 189900 OneFamames %>% select(Bldg_Type) %>% distinct() %>% pull()## [1] OneFam TwnhsE Twnhs Duplex TwoFmCon
## Levels: OneFam TwoFmCon Duplex Twnhs TwnhsEsimple_ames <- recipe(Sale_Price ~ Bldg_Type, data = ames) %>%
step_dummy(all_nominal_predictors()) %>%
prep()
simple_ames## Recipe
##
## Inputs:
##
## role #variables
## outcome 1
## predictor 1
##
## Training data contained 2930 data points and no missing data.
##
## Operations:
##
## Dummy variables from Bldg_Type [trained]simple_ames %>% bake(new_data = NULL) %>% head(5)## # A tibble: 5 × 5
## Sale_Price Bldg_Type_TwoFmC… Bldg_Type_Duplex Bldg_Type_Twnhs Bldg_Type_TwnhsE
## <int> <dbl> <dbl> <dbl> <dbl>
## 1 215000 0 0 0 0
## 2 105000 0 0 0 0
## 3 172000 0 0 0 0
## 4 244000 0 0 0 0
## 5 189900 0 0 0 0El proceso de dicotomización demanda que únicamente (n-1) categorías sean expresadas, mientras que la restante será considerada la categoría default o basal. Esta última categoría es la usada en el modelo cuando todas las demás se encuentran ausentes.
4.6.1.3 Codificación de datos cualitativos nuevos o faltantes
Una de las tareas de ingeniería de datos más comunes es el tratamiento de datos faltantes, datos no antes vistos y datos con poca frecuencia. El problema principal con estos casos es que los modelos no saben cómo relacionar estos eventos con futuras predicciones. Es conveniente realizar las transformaciones necesarias de tratamiento de estos datos antes de pasar a la etapa de modelado.
Por ejemplo:
step_unknown()cambia los valores perdidos en un nivel de factor “desconocido.”step_other()analiza las frecuencias de los niveles de los factores en el conjunto de datos y convierte los valores que ocurren con poca frecuencia a un nivel general de “otro,” con un umbral que se puede especificar.step_novel()puede asignar un nuevo nivel si anticipamos que se puede encontrar un nuevo factor en datos futuros.
Un buen ejemplo es el predictor de vecindad en nuestros datos. Aquí hay dos vecindarios que tienen menos de cinco propiedades.
ggplot(data = ames, aes(y = Neighborhood)) +
geom_bar() +
labs(y = NULL)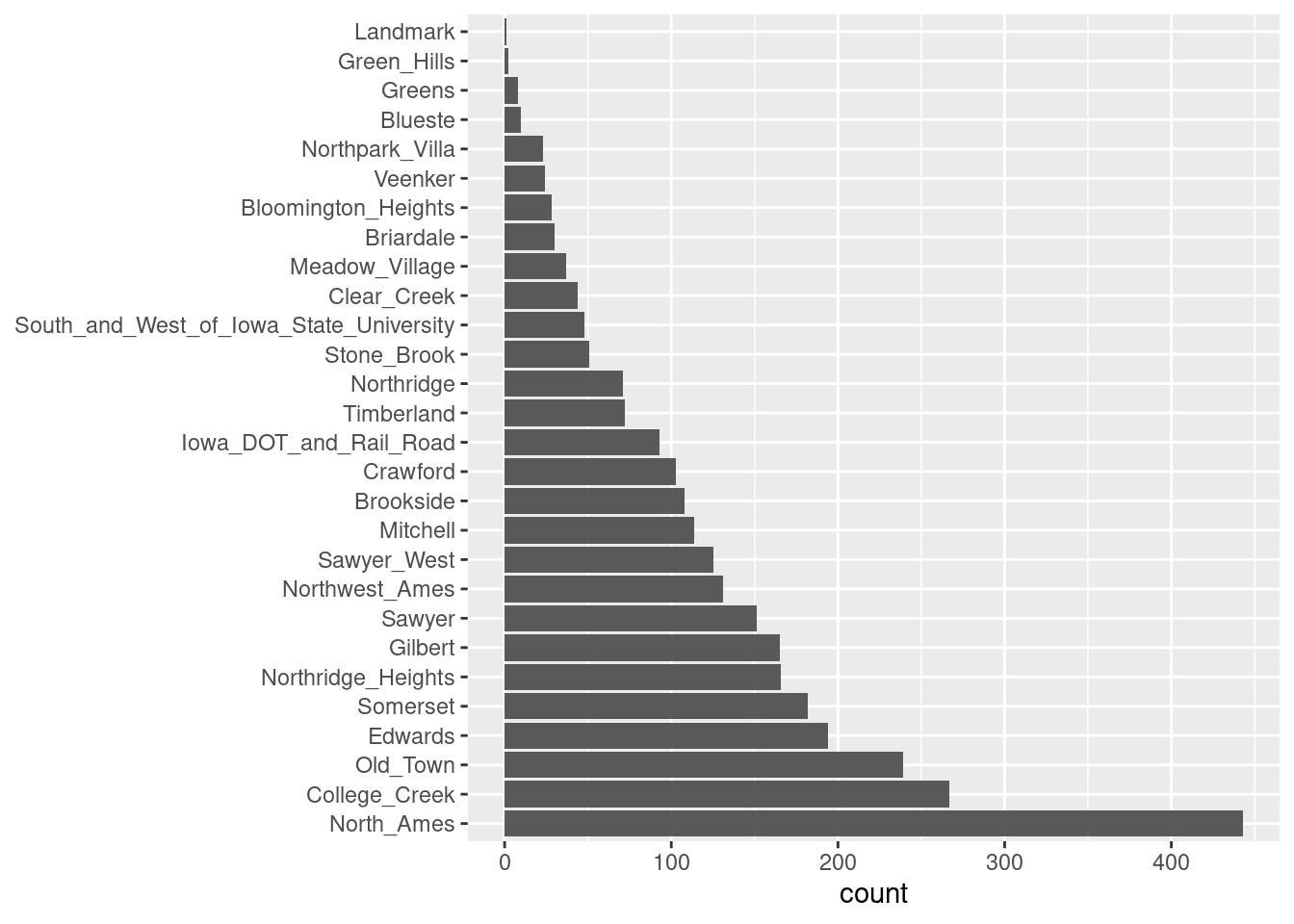
Para algunos modelos, puede resultar problemático tener variables dummy con una sola entrada distinta de cero en la columna. Como mínimo, es muy improbable que estas características sean importantes para un modelo.
Si agregamos step_other (Neighborhood, threshold = 0.01) a nuestra receta, el último \(1\%\) de los vecindarios se agrupará en un nuevo nivel llamado “otro,” esto atrapará a 8 vecindarios.
simple_ames <- recipe(
Sale_Price ~ Neighborhood + Gr_Liv_Area + Year_Built + Bldg_Type,
data = ames) %>%
step_other(Neighborhood, threshold = 0.01) %>%
prep()
ejemplo <- juice(simple_ames)
ggplot(ejemplo, aes(y = Neighborhood)) +
geom_bar() +
labs(y = NULL)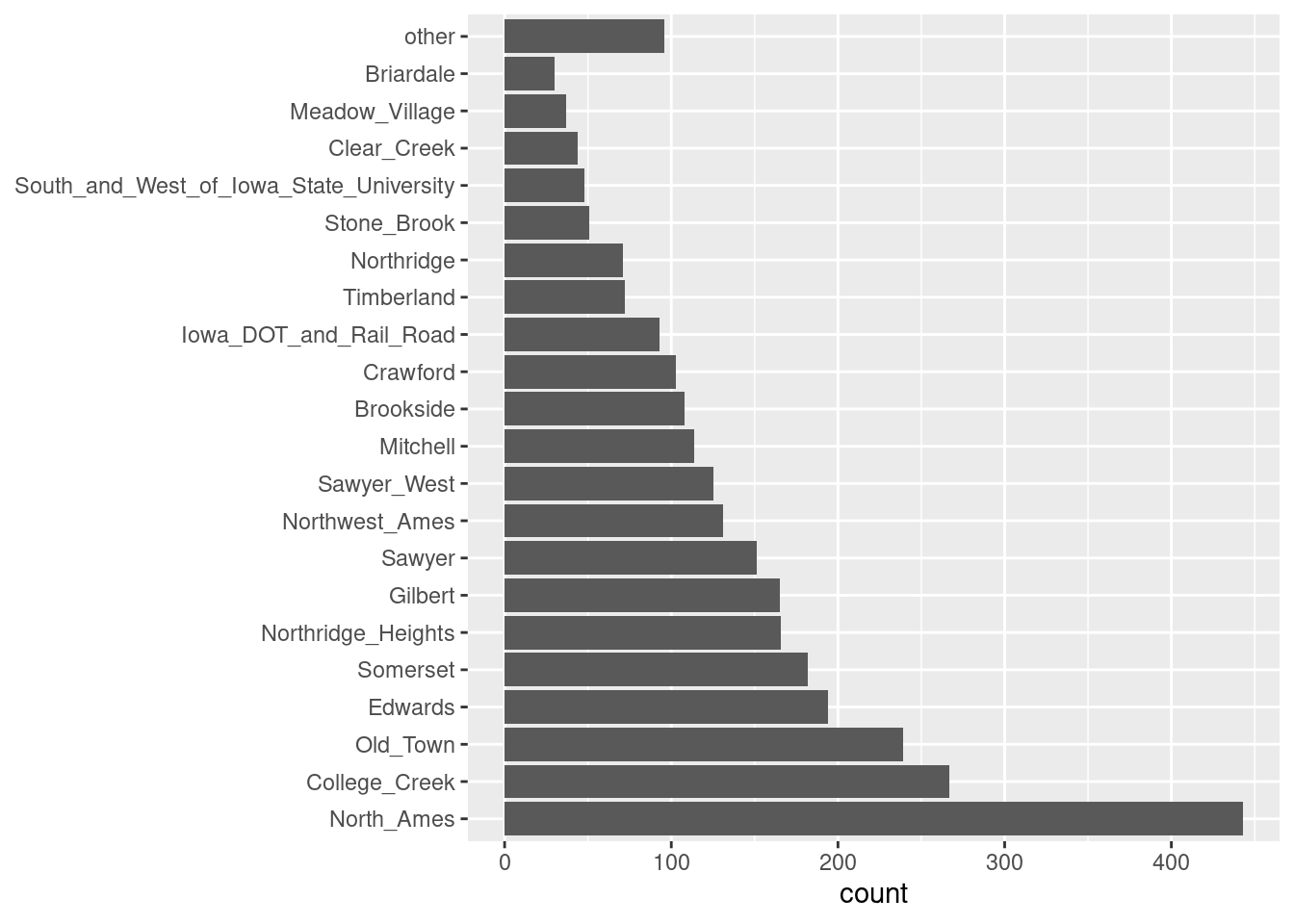
4.6.2 Imputaciones
La función step_unknown crea una categoría nombrada unknown, la cual sirve como reemplazo de datos categóricos faltantes, sin embargo, para imputar datos numéricos se requiere de otra estrategia. Las imputaciones o sustituciones más comunes son realizadas a través de medidas de tendencia central tales como la media y mediana. A continuación se muestra un ejemplo:
ames_na <- ames
ames_na[sample(nrow(ames), 5), c("Gr_Liv_Area", "Lot_Area")] <- NA
ames_na %>% filter(is.na(Gr_Liv_Area) | is.na(Lot_Area)) %>%
select(Sale_Price, Gr_Liv_Area, Lot_Area)## # A tibble: 5 × 3
## Sale_Price Gr_Liv_Area Lot_Area
## <int> <int> <int>
## 1 224000 NA NA
## 2 121500 NA NA
## 3 159434 NA NA
## 4 135960 NA NA
## 5 214000 NA NAsimple_ames <- recipe(Sale_Price ~ Gr_Liv_Area + Lot_Area, data = ames_na) %>%
step_impute_mean(Gr_Liv_Area) %>%
step_impute_median(Lot_Area) %>%
prep()
bake(simple_ames, new_data = ames_na) %>%
filter(is.na(Gr_Liv_Area) | is.na(Lot_Area))## # A tibble: 0 × 3
## # … with 3 variables: Gr_Liv_Area <int>, Lot_Area <int>, Sale_Price <int>Forzamos algunos renglones a que sean omitidos aleatoriamente. Posteriormente, estos valores son imputados mediante su media y mediana.
4.6.3 Agregar o modificar columnas
Quizá la transformación más usada sea la agregación o mutación de columnas existentes. Similar a la función mutate() de dplyr, la función step_mutate() se encarga de realizar esta tarea dentro de un pipeline o receta.
ejemplo <- recipe(
Sale_Price ~ Neighborhood + Gr_Liv_Area + Year_Built + Bldg_Type + Year_Remod_Add,
data = ames) %>%
step_mutate(
Sale_Price_Peso = Sale_Price * 19.87,
Last_Inversion = Year_Remod_Add - Year_Built
) %>%
step_arrange(desc(Last_Inversion)) %>%
prep()
ejemplo## Recipe
##
## Inputs:
##
## role #variables
## outcome 1
## predictor 5
##
## Training data contained 2930 data points and no missing data.
##
## Operations:
##
## Variable mutation for ~Sale_Price * 19.87, ~Year_Remod_Add - Yea... [trained]
## Row arrangement using ~desc(Last_Inversion) [trained]ejemplo %>% bake(new_data = NULL) %>%
select(Sale_Price, Sale_Price_Peso, Year_Remod_Add, Year_Built, Last_Inversion)## # A tibble: 2,930 × 5
## Sale_Price Sale_Price_Peso Year_Remod_Add Year_Built Last_Inversion
## <int> <dbl> <int> <int> <int>
## 1 131000 2602970 2007 1880 127
## 2 265979 5285003. 2003 1880 123
## 3 295000 5861650 2002 1880 122
## 4 94000 1867780 1996 1875 121
## 5 138000 2742060 2006 1890 116
## 6 122000 2424140 1987 1872 115
## 7 240000 4768800 2002 1890 112
## 8 119600 2376452 2006 1895 111
## 9 124000 2463880 1991 1880 111
## 10 100000 1987000 1995 1885 110
## # … with 2,920 more rowsEn este ejemplo se realiza la creación de una nueva variable y la modificación de una ya existente.
4.6.4 Interacciones
Los efectos de interacción involucran dos o más predictores. Tal efecto ocurre cuando un predictor tiene un efecto sobre el resultado que depende de uno o más predictores.
Numéricamente, un término de interacción entre predictores se codifica como su producto. Las interacciones solo se definen en términos de su efecto sobre el resultado y pueden ser combinaciones de diferentes tipos de datos (por ejemplo, numéricos, categóricos, etc.).
Después de explorar el conjunto de datos de Ames, podríamos encontrar que las pendientes de regresión para el área habitable bruta difieren para los diferentes tipos de edificios:
ggplot(ames, aes(x = Gr_Liv_Area, y = Sale_Price)) +
geom_point(alpha = .2) +
facet_wrap(~ Bldg_Type) +
geom_smooth(method = lm, formula = y ~ x, se = FALSE, col = "red") +
scale_x_log10() +
scale_y_log10() +
labs(x = "Gross Living Area", y = "Sale Price (USD)")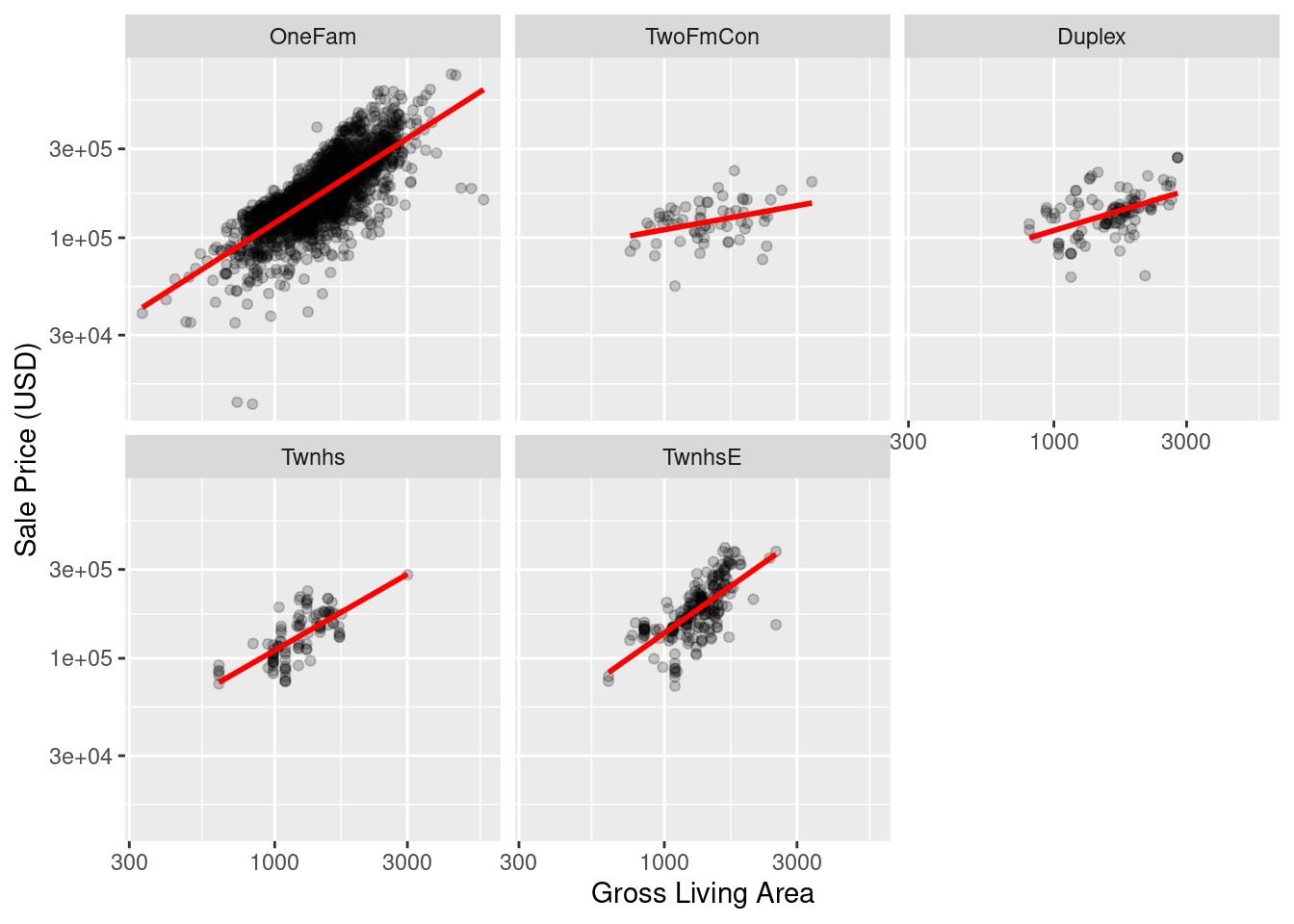
Con la receta actual, step_dummy() ya ha creado variables ficticias.
¿Cómo combinaríamos estos para una interacción? El paso adicional se vería como
step_interact(~ términos de interacción) donde los términos en el lado derecho de la tilde son las interacciones. Estos pueden incluir selectores, por lo que sería apropiado usar:
simple_ames <- recipe(Sale_Price ~ Neighborhood + Gr_Liv_Area + Year_Built + Bldg_Type,
data = ames) %>%
step_other(Neighborhood, threshold = 0.05) %>%
step_dummy(all_nominal_predictors()) %>%
step_interact( ~ Gr_Liv_Area:starts_with("Bldg_Type_") ) %>%
prep()
simple_ames %>% bake(new_data = NULL) %>% glimpse()## Rows: 2,930
## Columns: 19
## $ Gr_Liv_Area <int> 1656, 896, 1329, 2110, 1629, 1604, 13…
## $ Year_Built <int> 1960, 1961, 1958, 1968, 1997, 1998, 2…
## $ Sale_Price <int> 215000, 105000, 172000, 244000, 18990…
## $ Neighborhood_College_Creek <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
## $ Neighborhood_Old_Town <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
## $ Neighborhood_Edwards <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
## $ Neighborhood_Somerset <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
## $ Neighborhood_Northridge_Heights <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
## $ Neighborhood_Gilbert <dbl> 0, 0, 0, 0, 1, 1, 0, 0, 0, 1, 1, 1, 1…
## $ Neighborhood_Sawyer <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
## $ Neighborhood_other <dbl> 0, 0, 0, 0, 0, 0, 1, 1, 1, 0, 0, 0, 0…
## $ Bldg_Type_TwoFmCon <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
## $ Bldg_Type_Duplex <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
## $ Bldg_Type_Twnhs <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
## $ Bldg_Type_TwnhsE <dbl> 0, 0, 0, 0, 0, 0, 1, 1, 1, 0, 0, 0, 0…
## $ Gr_Liv_Area_x_Bldg_Type_TwoFmCon <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
## $ Gr_Liv_Area_x_Bldg_Type_Duplex <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
## $ Gr_Liv_Area_x_Bldg_Type_Twnhs <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
## $ Gr_Liv_Area_x_Bldg_Type_TwnhsE <dbl> 0, 0, 0, 0, 0, 0, 1338, 1280, 1616, 0…Se pueden especificar interacciones adicionales en esta fórmula separándolas con el signo \(*\).
4.6.5 Transformaciones generales
Reflejando las operaciones originales de dplyr, los siguientes pasos se pueden usar para realizar una variedad de operaciones básicas a los datos.
step_select(): Selecciona un subconjunto de variables específicas en el conjunto de datos.step_mutate(): Crea una nueva variable o modifica una existente usandodplyr::mutate().step_mutate_at(): Lee una especificación de un paso de receta que modificará las variables seleccionadas usando una función común a través dedplyr::mutate_at().step_filter(): Crea una especificación de un paso de receta que eliminará filas usandodplyr::filter().step_arrange(): Ordena el conjunto de datos de acuerdo con una o más variables.step_rm(): Crea una especificación de un paso de receta que eliminará las variables según su nombre, tipo o función.step_nzv(): Realiza una selección de variables eliminando todas aquellas cuya varianza se encuentre cercana a cero.step_naomit(): Elimina todos los renglones que tengan alguna variable con valores perdidos.step_normalize(): Centra y escala las variables numéricas especificadas, generando una transformación a una distribución normal estándar.step_range(): Transforma el rango de un conjunto de variables numéricas al especificado.step_interact(): Crea un nuevo conjunto de variables basadas en la interacción entre dos variables.step_ratio(): Crea una nueva variable a partir del cociente entre dos variables.all_predictors(): Selecciona a todos los predictores del conjunto de entrenamineto para aplicarles alguna de las funciones mencionadas.all_numeric_predictors(): Selecciona a todos los predictores numéricos del conjunto de entrenamineto para aplicarles alguna de las funciones mencionadas.all_nominal_predictors(): Selecciona a todos los predictores nominales del conjunto de entrenamineto para aplicarles alguna de las funciones mencionadas.
La guía completa de las familia de funciones step puede consultarse en la documentación oficial
4.7 Partición de datos

Cuando hay una gran cantidad de datos disponibles, una estrategia inteligente es asignar subconjuntos específicos de datos para diferentes tareas, en lugar de asignar la mayor cantidad posible solo a la estimación de los parámetros del modelo.
Si el conjunto inicial de datos no es lo suficientemente grande, habrá cierta superposición de cómo y cuándo se asignan nuestros datos, y es importante contar con una metodología sólida para la partición de datos.
4.7.1 Métodos comunes para particionar datos
El enfoque principal para la validación del modelo es dividir el conjunto de datos existente en dos conjuntos distintos:
Entrenamiento: Este conjunto suele contener la mayoría de los datos, los cuales sirven para la construcción de modelos donde se pueden ajustar diferentes modelos, se investigan estrategias de ingeniería de características, etc.
La mayor parte del proceso de modelado se utiliza este conjunto.
Prueba: La otra parte de las observaciones se coloca en este conjunto. Estos datos se mantienen en reserva hasta que se elijan uno o dos modelos como los de mejor rendimiento.
El conjunto de prueba se utiliza como árbitro final para determinar la eficiencia del modelo, por lo que es fundamental mirar el conjunto de prueba una sola vez.
Supongamos que asignamos el \(80\%\) de los datos al conjunto de entrenamiento y el \(20\%\) restante a las pruebas. El método más común es utilizar un muestreo aleatorio simple.
El paquete rsample tiene herramientas para realizar divisiones de datos como esta;
la función initial_split() fue creada para este propósito.
library(tidymodels)
tidymodels_prefer()
# Fijar un número aleatorio con para que los resultados puedan ser reproducibles
set.seed(123)
# Partición 80/20 de los datos
ames_split <- initial_split(ames, prop = 0.80)
ames_split## <Analysis/Assess/Total>
## <2344/586/2930>La información impresa denota la cantidad de datos en el conjunto de entrenamiento \((n = 2,344)\), la cantidad en el conjunto de prueba \((n = 586)\) y el tamaño del grupo original de muestras \((n = 2,930)\).
El objeto ames_split es un objeto rsplit y solo contiene la información de partición; para obtener los conjuntos de datos resultantes, aplicamos dos funciones más:
ames_train <- training(ames_split)
ames_test <- testing(ames_split)
dim(ames_train)## [1] 2344 74El muestreo aleatorio simple es apropiado en muchos casos, pero hay excepciones.
Cuando hay un desbalance de clases en los problemas de clasificación, el uso de una muestra aleatoria simple puede asignar al azar estas muestras poco frecuentes de manera desproporcionada al conjunto de entrenamiento o prueba.
Para evitar esto, se puede utilizar un muestreo estratificado. La división de entrenamiento/prueba se lleva a cabo por separado dentro de cada clase y luego estas submuestras se combinan en el conjunto general de entrenamiento y prueba.
Para los problemas de regresión, los datos de los resultados se pueden agrupar artificialmente en cuartiles y luego realizar un muestreo estratificado cuatro veces por separado. Este es un método eficaz para mantener similares las distribuciones del resultado entre el conjunto de entrenamiento y prueba.
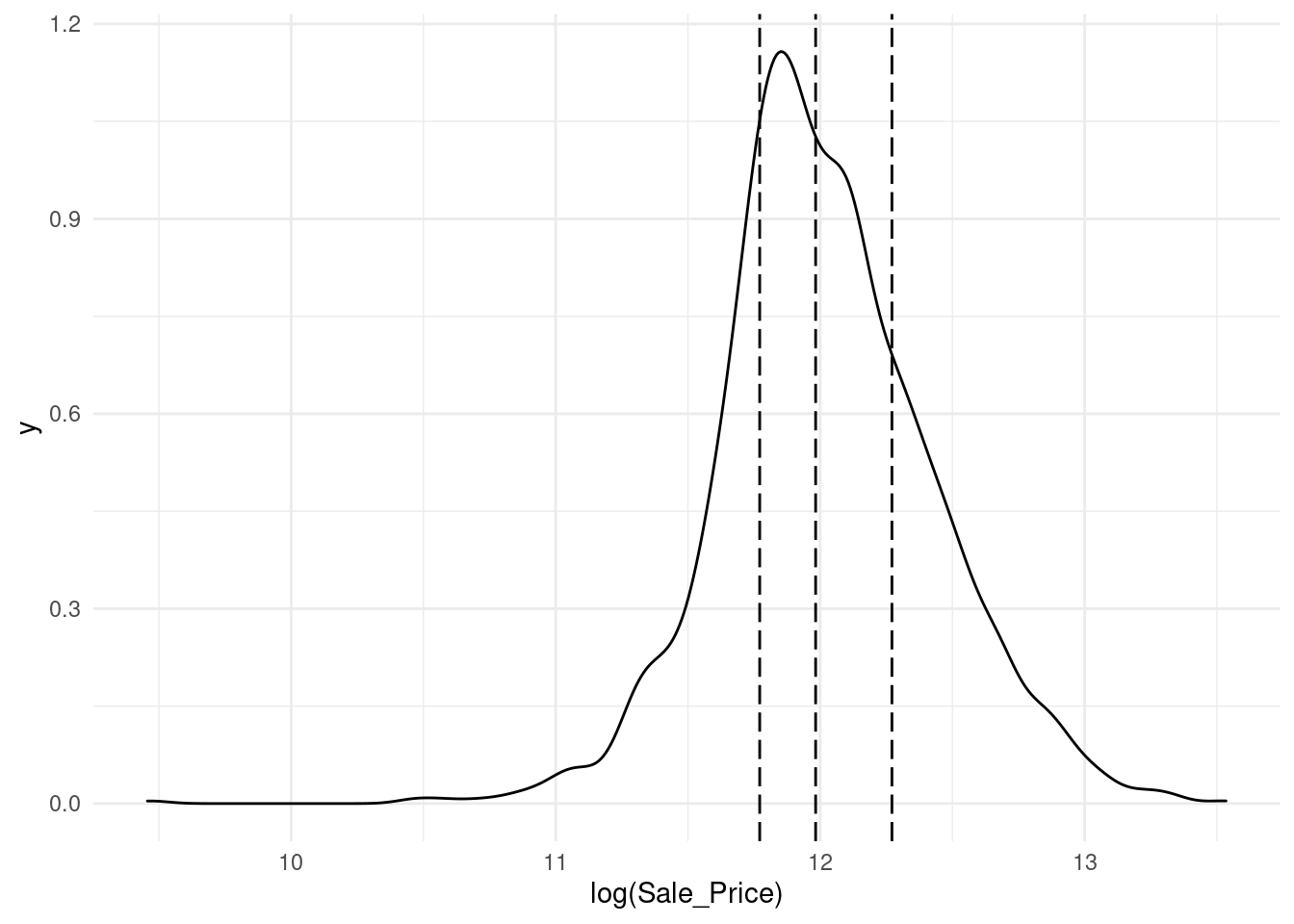
Observamos que la distribución del precio de venta está sesgada a la derecha. Las casas más caras no estarían bien representadas en el conjunto de entrenamiento con una simple partición; esto aumentaría el riesgo de que nuestro modelo sea ineficaz para predecir el precio de dichas propiedades.
Las líneas verticales punteadas indican los cuatro cuartiles para estos datos. Una muestra aleatoria estratificada llevaría a cabo la división 80/20 dentro de cada uno de estos subconjuntos de datos y luego combinaría los resultados. En rsample, esto se logra usando el argumento de estratos:
set.seed(123)
ames_split <- initial_split(ames, prop = 0.80, strata = Sale_Price)
ames_train <- training(ames_split)
ames_test <- testing(ames_split)Hay muy pocas desventajas en el uso de muestreo estratificado.
Un caso es cuando los datos tienen un componente de tiempo, como los datos de series de tiempo. Aquí, es más común utilizar los datos más recientes como conjunto de prueba.
El paquete rsample contiene una función llamada initial_time_split()
que es muy similar a initial_split(). En lugar de usar un muestreo aleatorio, el argumento prop
denota qué proporción de la primera parte de los datos debe usarse como conjunto de entrenamiento;
la función asume que los datos se han clasificado previamente en un orden apropiado.
4.7.2 ¿Qué proporción debería ser usada?
No hay un porcentaje de división óptimo para el conjunto de entrenamiento y prueba. Muy pocos datos en el conjunto de entrenamiento obstaculizan la capacidad del modelo para encontrar estimaciones de parámetros adecuadas y muy pocos datos en el conjunto de prueba reducen la calidad de las estimaciones de rendimiento.
Se debe elegir un porcentaje que cumpla con los objetivos de nuestro proyecto con consideraciones que incluyen:
- Costo computacional en el entrenamiento del modelo.
- Costo computacional en la evaluación del modelo.
- Representatividad del conjunto de formación.
- Representatividad del conjunto de pruebas.
Los porcentajes de división más comunes comunes son:
- Entrenamiento: \(80\%\), Prueba: \(20\%\)
- Entrenamiento: \(67\%\), Prueba: \(33\%\)
- Entrenamiento: \(50\%\), Prueba: \(50\%\)
4.7.3 Conjunto de validación
El conjunto de validación se definió originalmente cuando los investigadores se dieron cuenta de que medir el rendimiento del conjunto de entrenamiento conducía a resultados que eran demasiado optimistas.
Esto llevó a modelos que se sobreajustaban, lo que significa que se desempeñaron muy bien en el conjunto de entrenamiento pero mal en el conjunto de prueba.
Para combatir este problema, se retuvo un pequeño conjunto de datos de validación y se utilizó para medir el rendimiento del modelo mientras este está siendo entrenado. Una vez que la tasa de error del conjunto de validación comenzara a aumentar, la capacitación se detendría.
En otras palabras, el conjunto de validación es un medio para tener una idea aproximada de qué tan bien se desempeñó el modelo antes del conjunto de prueba.
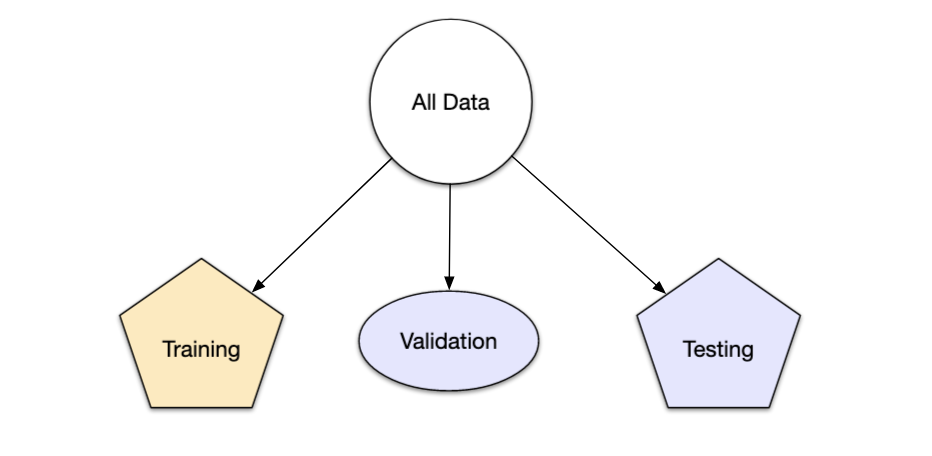
Los conjuntos de validación se utilizan a menudo cuando el conjunto de datos original es muy grande. En este caso, una sola partición grande puede ser adecuada para caracterizar el rendimiento del modelo sin tener que realizar múltiples iteraciones de remuestreo.
Con rsample, un conjunto de validación es como cualquier otro objeto de remuestreo; este tipo es diferente solo en que tiene una sola iteración
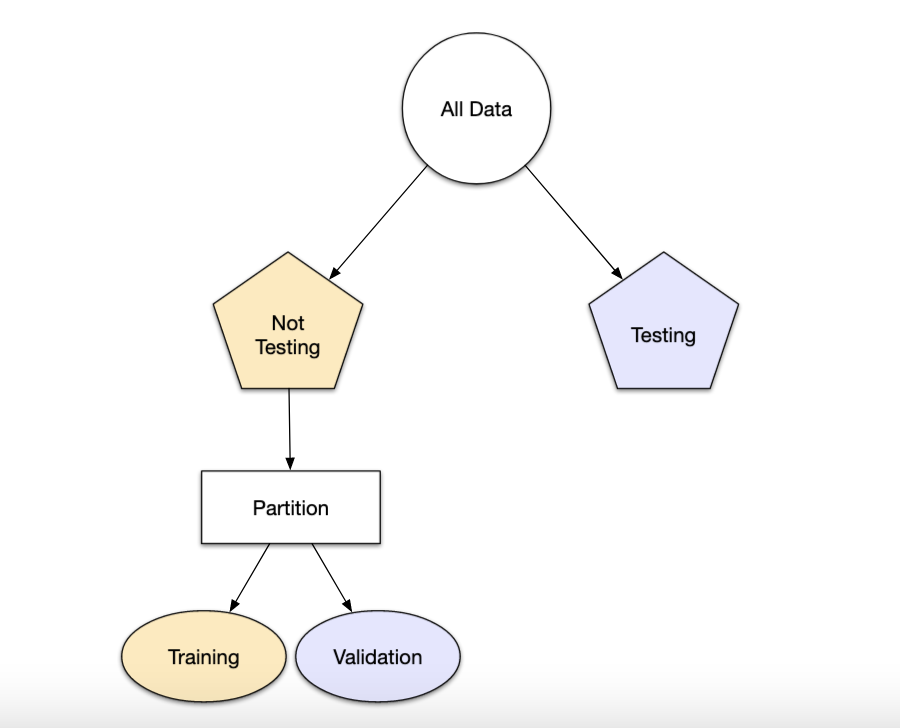
set.seed(12)
val_set <- validation_split(ames_train, prop = 3/4, strata = NULL)
val_set #val_set contiene el conjunto de entrenamiento y validación.## # Validation Set Split (0.75/0.25)
## # A tibble: 1 × 2
## splits id
## <list> <chr>
## 1 <split [1756/586]> validationEsta función regresa una columna para los objetos de división de datos y una columna llamada id que tiene una cadena de caracteres con el identificador de remuestreo.
El argumento de estratos hace que el muestreo aleatorio se lleve a cabo dentro de la variable de estratificación. Esto puede ayudar a garantizar que el número de datos en los datos del análisis sea equivalente a las proporciones del conjunto de datos original. (Los estratos inferiores al 10% del total se agrupan).
Otra opción de muestreo bastante común es la realizada mediante múltiples submuestras de los datos originales.

Diversos métodos se revisarán a lo largo del curso.
4.7.4 Leave-one-out cross-validation
La validación cruzada es una manera de predecir el ajuste de un modelo a un hipotético conjunto de datos de prueba cuando no disponemos del conjunto explícito de datos de prueba.
El método LOOCV en un método iterativo que se inicia empleando como conjunto de entrenamiento todas las observaciones disponibles excepto una, que se excluye para emplearla como validación.
Si se emplea una única observación para calcular el error, este varía mucho dependiendo de qué observación se haya seleccionado. Para evitarlo, el proceso se repite tantas veces como observaciones disponibles se tengan, excluyendo en cada iteración una observación distinta, ajustando el modelo con el resto y calculando el error con dicha observación.
Finalmente, el error estimado por el es el promedio de todos lo \(i\) errores calculados.
La principal desventaja de este método es su costo computacional. El proceso requiere que el modelo sea reajustado y validado tantas veces como observaciones disponibles se tengan lo que en algunos casos puede ser muy complicado.
rsample contiene la función loo_cv().
set.seed(55)
ames_loo <- loo_cv(ames_train)
ames_loo## # Leave-one-out cross-validation
## # A tibble: 2,342 × 2
## splits id
## <list> <chr>
## 1 <split [2341/1]> Resample1
## 2 <split [2341/1]> Resample2
## 3 <split [2341/1]> Resample3
## 4 <split [2341/1]> Resample4
## 5 <split [2341/1]> Resample5
## 6 <split [2341/1]> Resample6
## 7 <split [2341/1]> Resample7
## 8 <split [2341/1]> Resample8
## 9 <split [2341/1]> Resample9
## 10 <split [2341/1]> Resample10
## # … with 2,332 more rows4.7.4.1 Cálculo del error
En la validación cruzada dejando uno fuera se realizan tantas iteraciones como muestras \((N)\) tenga el conjunto de datos. De forma que para cada una de las \(N\) iteraciones se realiza un cálculo de error.
El resultado final se obtiene realizando la media de los \(N\) errores obtenidos, según la fórmula:
\[E = \frac{1}{N}\sum_{i = 1}^N E_i\]
4.7.5 V Fold Cross Validation
En la validación cruzada de V iteraciones (V Fold Cross Validation) los datos de muestra se dividen en V subconjuntos. Uno de los subconjuntos se utiliza como datos de prueba y el resto \((V-1)\) como datos de entrenamiento. El proceso de validación cruzada es repetido durante \(v\) iteraciones, con cada uno de los posibles subconjuntos de datos de prueba.
Finalmente se obtiene el promedio de los rendimientos de cada iteración para obtener un único resultado. Lo más común es utilizar la validación cruzada de 10 iteraciones.

Este método de validación cruzada se utiliza principalmente para:
Estimar el error cuando nuestro conjunto de prueba es muy pequeño. Es decir, se tiene la misma confuguración de parámetos y solamente cambia el conjunto de prueba y validación.
Encontrar lo mejores hiperparámetros que ajusten mejor el modelo. Es decir, en cada bloque se tiene una configuración de hiperparámetros distinto y se seleccionará aquellos hiperparámetros que hayan producido el error más pequeño.

En la función vfold_cv() la entrada principal es el conjunto de entrenamiento,
así como el número de bloques:
set.seed(55)
ames_folds <- vfold_cv(ames_train, v = 10)
ames_folds## # 10-fold cross-validation
## # A tibble: 10 × 2
## splits id
## <list> <chr>
## 1 <split [2107/235]> Fold01
## 2 <split [2107/235]> Fold02
## 3 <split [2108/234]> Fold03
## 4 <split [2108/234]> Fold04
## 5 <split [2108/234]> Fold05
## 6 <split [2108/234]> Fold06
## 7 <split [2108/234]> Fold07
## 8 <split [2108/234]> Fold08
## 9 <split [2108/234]> Fold09
## 10 <split [2108/234]> Fold10La columna denominada splits contiene la información sobre cómo dividir los datos
(similar al objeto utilizado para crear la partición inicial de entrenamiento / prueba).
Si bien cada fila de divisiones tiene una copia incrustada de todo el conjunto de entrenamiento, R es lo suficientemente inteligente como para no hacer copias de los datos en la memoria.
El método de impresión dentro del tibble muestra la frecuencia de cada uno: [2K / 230] indica que aproximadamente dos mil muestras están en el conjunto de análisis y 230 están en ese conjunto de evaluación en particular.
Estos objetos rsample también contienen siempre una columna de caracteres llamada id que etiqueta la partición. Algunos métodos de remuestreo requieren varios campos de identificación.
Para recuperar manualmente los datos particionados, las funciones de analysis()
y assessment() devuelven los de datos de análisis y evaluación respectivamente.
# Primer bloque
ames_folds$splits[[1]] %>%
analysis() %>% # O assessment()
head(7)## # A tibble: 7 × 74
## MS_SubClass MS_Zoning Lot_Frontage Lot_Area Street Alley Lot_Shape
## <fct> <fct> <dbl> <int> <fct> <fct> <fct>
## 1 One_Story_1946_and_New… Resident… 70 8400 Pave No_A… Regular
## 2 Two_Story_PUD_1946_and… Resident… 21 1680 Pave No_A… Regular
## 3 Two_Story_PUD_1946_and… Resident… 21 1680 Pave No_A… Regular
## 4 Two_Story_PUD_1946_and… Resident… 21 1680 Pave No_A… Regular
## 5 One_Story_PUD_1946_and… Resident… 53 4043 Pave No_A… Regular
## 6 One_Story_PUD_1946_and… Resident… 24 2280 Pave No_A… Regular
## 7 One_Story_PUD_1946_and… Resident… 50 7175 Pave No_A… Regular
## # … with 67 more variables: Land_Contour <fct>, Utilities <fct>,
## # Lot_Config <fct>, Land_Slope <fct>, Neighborhood <fct>, Condition_1 <fct>,
## # Condition_2 <fct>, Bldg_Type <fct>, House_Style <fct>, Overall_Cond <fct>,
## # Year_Built <int>, Year_Remod_Add <int>, Roof_Style <fct>, Roof_Matl <fct>,
## # Exterior_1st <fct>, Exterior_2nd <fct>, Mas_Vnr_Type <fct>,
## # Mas_Vnr_Area <dbl>, Exter_Cond <fct>, Foundation <fct>, Bsmt_Cond <fct>,
## # Bsmt_Exposure <fct>, BsmtFin_Type_1 <fct>, BsmtFin_SF_1 <dbl>, …4.7.6 Medidas de ajuste
Las medidas de ajuste obtenidas pueden ser utilizadas para estimar cualquier medida cuantitativa de ajuste apropiada para los datos y el modelo.
En un modelo basado en clasificación binaria, para resumir el ajuste del modelo se pueden usar las medidas:
- Tasa de error de clasificación (Accuracy)
- Precisión
- Sensibilidad o coertura (Recall)
- Especificidad
Cuando el valor a predecir se distribuye de forma continua se puede calcular el error utilizando medidas como:
- Error porcentual absoluto medio (MAPE)
- Error absoluto medio (MAE)
- Error cuadrático medio (MSE)
- Raíz del error cuadrático medio (RMSE)
- Raíz del error logarítmico cuadrático medio (RMLSE)
- \(R^2\) (Coeficiente de determinación)
- \(R^2_a\) (Coeficiente de determinación ajustado)
4.7.6.1 Cálculo del error
En cada una de las \(v\) iteraciones de este tipo de validación se realiza un cálculo de error. El resultado final lo obtenemos a partir de realizar la media de los \(V\) valores de errores obtenidos, según la fórmula:
\[E = \frac{1}{V}\sum_{i = 1}^vE_i\]
4.7.7 Validación cruzada para series de tiempo
En este procedimiento, hay una serie de conjuntos de prueba, cada uno de los cuales consta de una única observación. El conjunto de entrenamiento correspondiente consta solo de observaciones que ocurrieron antes de la observación que forma el conjunto de prueba. Por lo tanto, no se pueden utilizar observaciones futuras para construir el pronóstico.
El siguiente diagrama ilustra la serie de conjuntos de entrenamiento y prueba, donde las observaciones azules forman los conjuntos de entrenamiento y las observaciones rojas forman los conjuntos de prueba.

La precisión del pronóstico se calcula promediando los conjuntos de prueba. Este procedimiento a veces se conoce como “evaluación en un origen de pronóstico continuo” porque el “origen” en el que se basa el pronóstico avanza en el tiempo.
Con los pronósticos de series de tiempo, los pronósticos de un paso pueden no ser tan relevantes como los pronósticos de varios pasos. En este caso, el procedimiento de validación cruzada basado en un origen de pronóstico continuo se puede modificar para permitir el uso de errores de varios pasos.
Suponga que estamos interesados en modelos que producen buenos pronósticos de 4 pasos por delante. Entonces el diagrama correspondiente se muestra a continuación.
 La validación cruzada de series de tiempo se implementa con la función
La validación cruzada de series de tiempo se implementa con la función tsCV()
del paquete forecast.